用TwoSampleMR包做孟德尔随机化分析
如题。
一、为什么要做孟德尔随机化
一言以蔽之:相关性 ≠ 因果性。
在研究中我们常常观察到一些相关性,例如人群中某个基因与某种疾病的发病率正相关。然而这种相关性并不能解释基因和疾病的关联,有可能是基因产物造成了这种疾病(真因果),有可能是这个基因的连锁基因的产物造成了疾病,甚至有可能是疾病的病因(例如逆转录病毒的感染)造成了这个基因的异常……
相关性说明不了因果性,因为存在第三种或更多因素,而造成相关性的情况也有很多种。简单概括一下,下面这些情况都会造成相关性:
- 因果相关:A导致B,B导致C (A→B→C)
- 混杂相关:A导致B跟C,造成B和C相关 (A→B,A→C)
- 对撞相关:A跟B共同导致C,控制C后A跟B相关 (A→C,B→C,(C):A→B)
- 随机相关:高维度数据中碰巧两个变量表现出了相关性
孟德尔随机化方法是一种因果分析方法,灵感来源于经济学研究中的工具变量分析。它可以区分上述这些情况,并正确估计或探索变量间的关系。
二、孟德尔随机化的原理
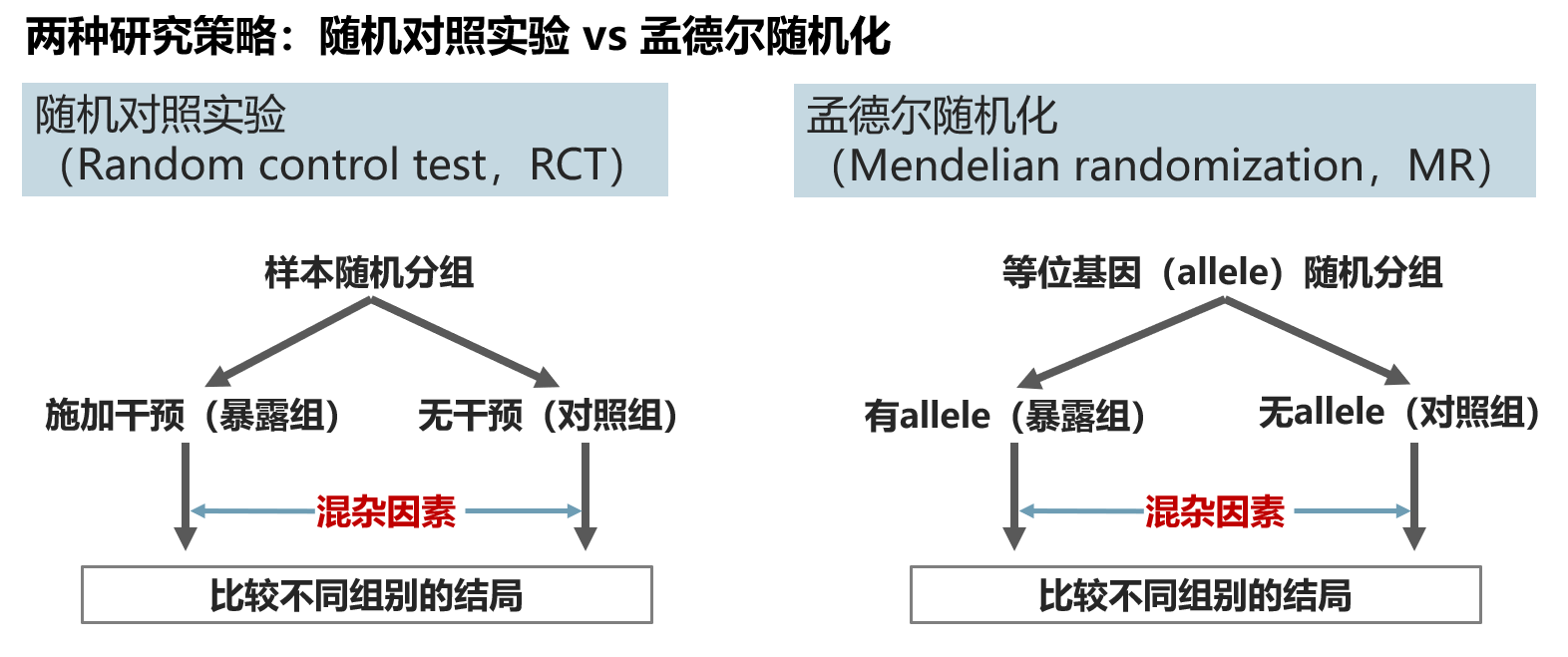
如上图。
某种意义上说,随机对照实验(RCT)更能揭示因果关系。然而RCT需要实验,成本很高。
孟德尔随机化(MR)的思路与CRT一致,但“随机分组”的过程由孟德尔分离定律所代替,因此无需实验,可以仅凭观察数据得出结论,实验成本更低,并且保证了最终结论的准确性。
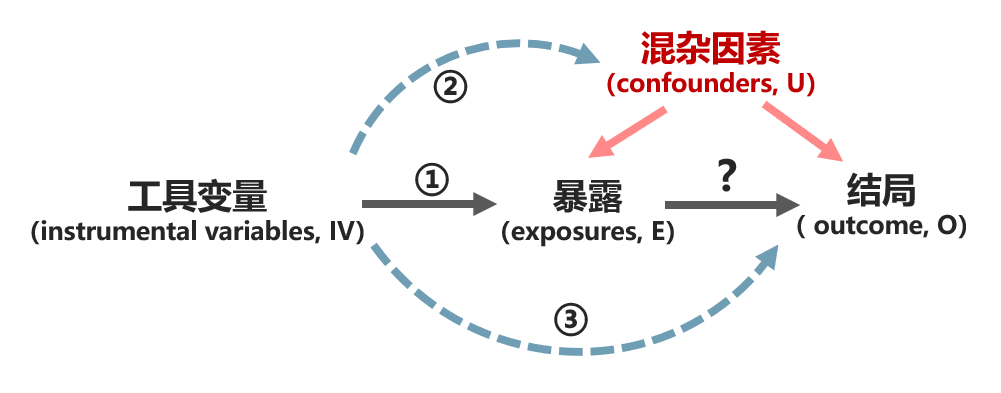
孟德尔随机化主要用于确定暴露因素(E)和结局(O)之间的因果关系。为了排除混杂因素的干扰,需要引入工具变量(IV),在此次是与且仅与暴露相关的一组遗传变异。作为工具变量的遗传变异满足下面三个核心假设:
- 1、关联性假设:工具变量(IV)与暴露(E)存在关联(图中①)
- 2、独立性假设:工具变量(IV)与混杂因素(U)不存在关联(图中②)
- 3、排他性假设:工具变量(IV)与结局(O)没有任何关联(图中③),除非通过暴露(E)(图中IV→E→O)
例如,我们想要研究咖啡因摄入如何影响肾结石发病率,可以这样选择:
- 工具变量(IV):通过GWAS找到的一组与咖啡因摄入有关的SNP
- 暴露(exposure):咖啡因摄入水平
- 结局(outcome):肾结石发病率
有多种方法可以用于计算MR,这些方法包括Wald ratio方法、逆方差加权法(IVW)、MR-Egger法等。不同方法有不同的适用情景,如果工具变量数量太少(只有一两个),那么Wald ratio方法会很合适。当有多个工具变量时,我们可以对每个工具变量各自计算Wald ratio,最后进行加权求和,这就是逆方差加权法(IVW)和MR-Egger法的原理。
有多种工具可以方便我们计算MR,下面我们只介绍一种:TwoSampleMR
三、TwoSampleMR包的安装与使用
TwoSampleMR包是一个用R语言编写的MR计算工具,其连接到了IEU GWAS database的后端,因此可以很方便的进行工具变量选取。
TwoSampleMR包暂时没有被CRAN收录,但作者提供了GitHub安装方法。在R语言的交互环境中输入下面的指令即可进行安装:
1 | install.packages("remotes") |
如果网络不太好,也可也从上述GitHub存储库中的release页面上直接下载.tar.gz格式的源码包,然后在R语言交互式环境中使用下面的指令进行源码包安装:
1 | install.packages("TwoSampleMR-0.5.10.tar.gz",repo=NULL,type="source") |
此外,TwoSampleMR 包依赖于 RadialMR 包( https://github.com/WSpiller/RadialMR ),因此如果上述安装方法出现问题,可以尝试先安装RadialMR包:
1 | remotes::install_github("WSpiller/RadialMR") |
然后再安装TwoSampleMR。
使用方法也很简单。下面这个例子来自TwoSampleMR的官方文档,研究的是身体质量指数(BMI)与冠心病(CDH)风险的因果关系。其中,BMI是暴露,而CDH是结局,我们要寻找一系列与BMI有关的工具变量SNP对这种因果关系进行检验。示例代码如下:
1 | library(TwoSampleMR) |
输出:
1 | id.exposure id.outcome outcome |
从输出可以看出,对于BMI这一暴露因素和冠心病(CHD)这一结局,使用MR-Egger方法给出的β值为0.5024935,显著大于0($p=8.01259^{-4}<0.05$ ),使用IVW方法给出的β值 0.4459091,同样显著大于0($p=4.03202^{-14}<0.05$ ),这说明BMI对CHD有比较强的正向因果效应。
最后,我们也可也画一个图,展示一下上面的结果:
1 | res <- mr(dat) |
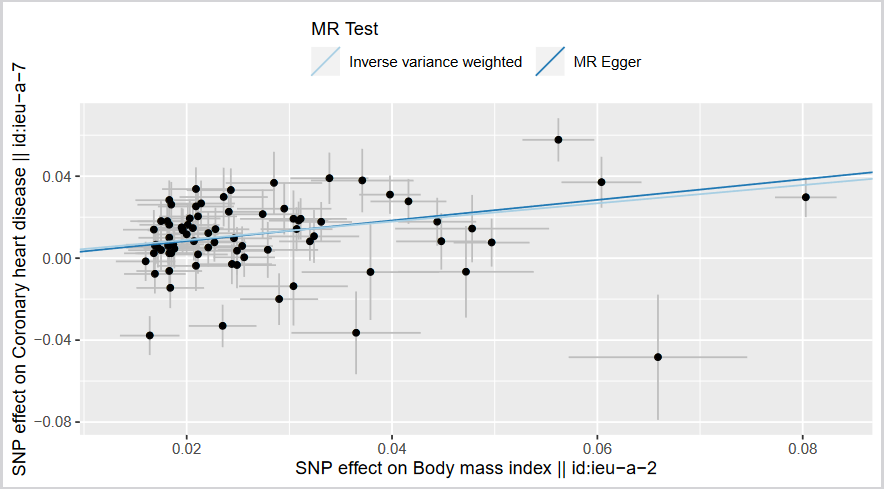
图中每个点代表一个工具变量对暴露的效应和对结局的效应。加权线性回归拟合出的直线斜率大于1,这说明BMI对CHD有正向因果效应。
参考文献
- https://mrcieu.github.io/TwoSampleMR/articles/introduction.html
- https://github.com/mrcieu/ieugwasr
- http://gwas-api.mrcieu.ac.uk/docs/
- https://github.com/MRCIEU/TwoSampleMR
- https://yufree.github.io/sciguide/thought.html#因果
- Sanderson, E., Glymour, M.M., Holmes, M.V. et al. Mendelian randomization. Nat Rev Methods Primers 2, 6 (2022).
- https://zhuanlan.zhihu.com/p/106666439