单细胞分析方法概要
本文内容来自《计算生物学原理与方法》2025级助教课PPT。
近期可能会更新一些单细胞分析的内容,本文则将作为这个系列的一个引子。
其他可参考的文章(博客的先前文章):
背景:为什么需要单细胞测序

单细胞测序能够发现细胞之间的异质性。更能够发现一些bulk-seq水平上无法发现的信息。
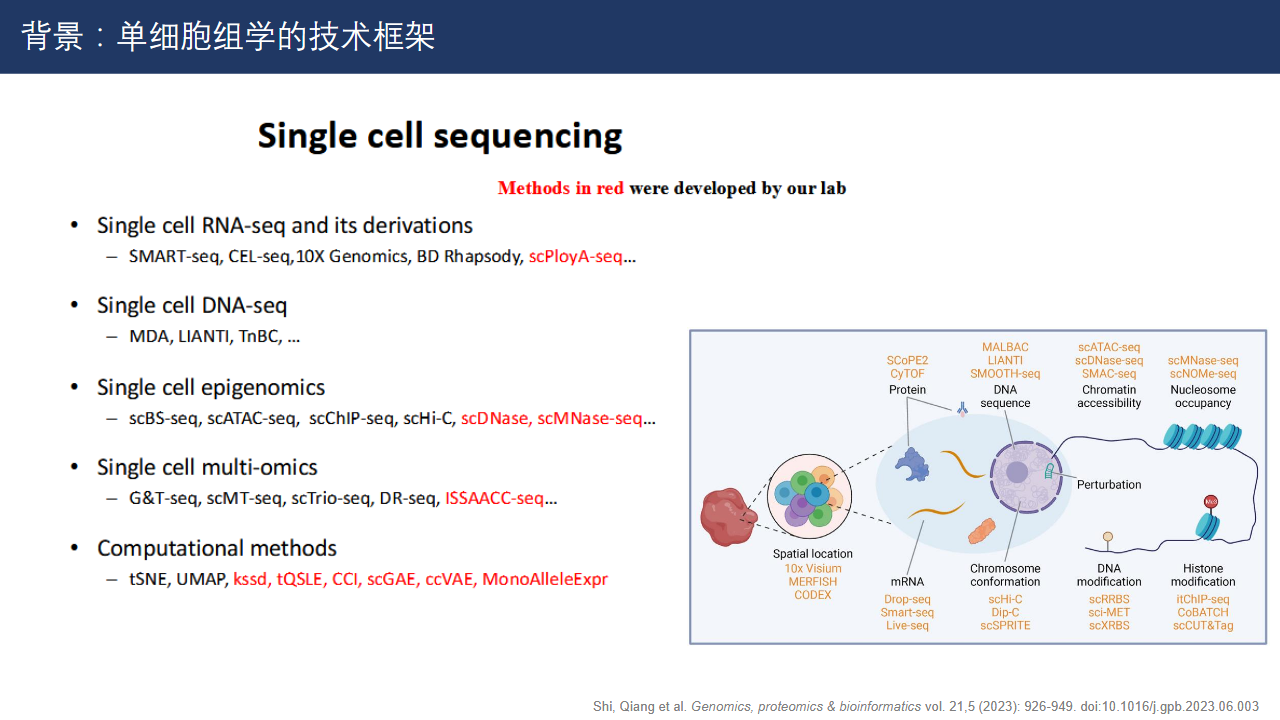
目前人们已经提出了许多测序技术框架。
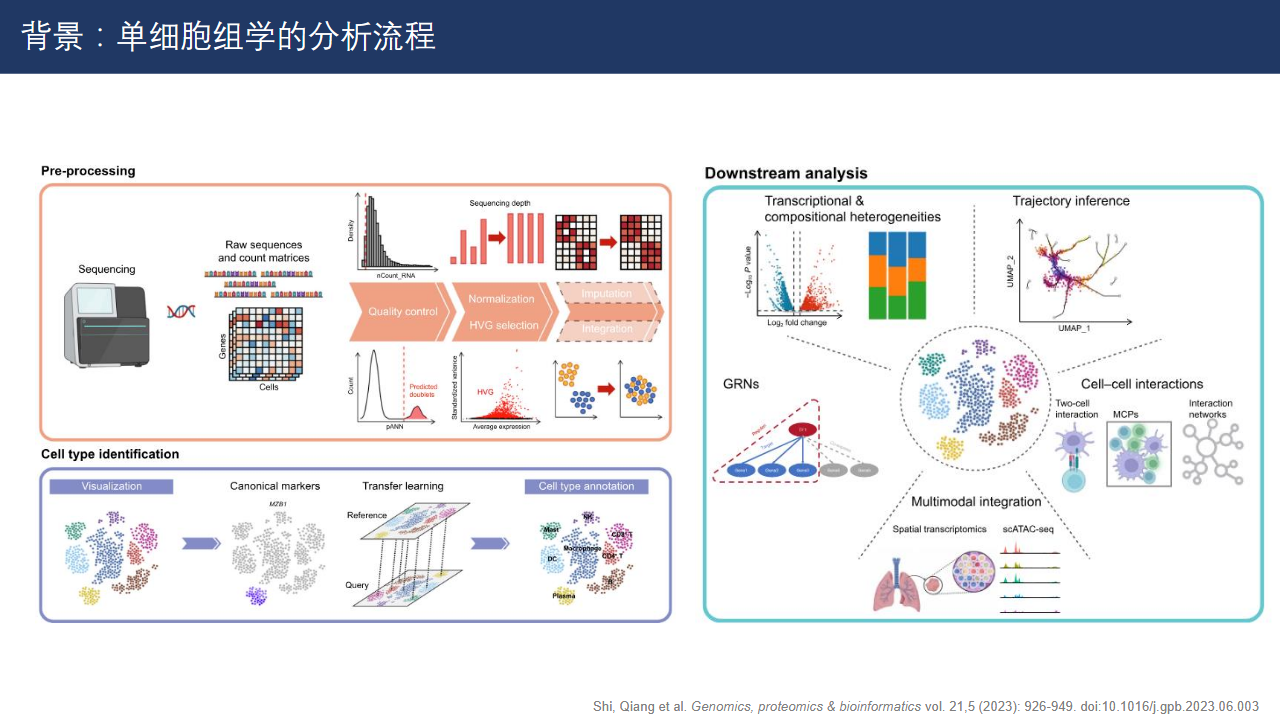
单细胞分析可以主要分为三大步骤:预处理(建库、上机测序、数据过滤和定量等)、细胞类型鉴定、下游分析。
单细胞分析的流程
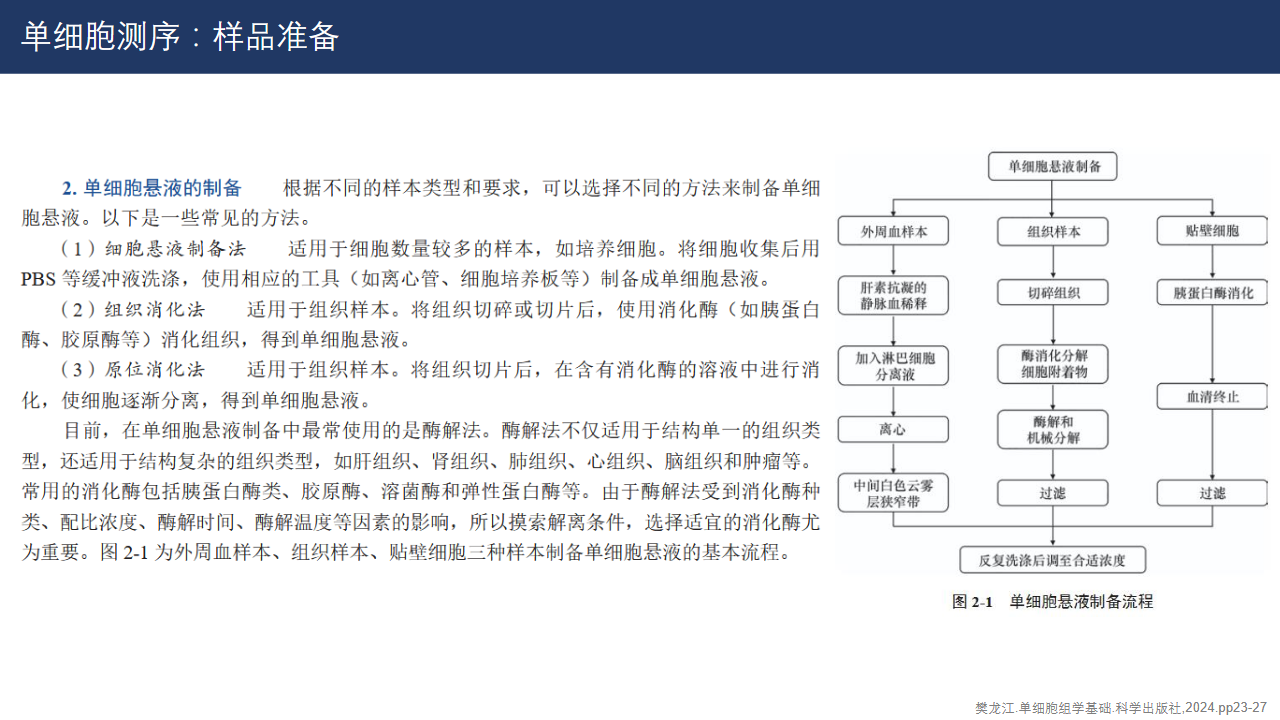
最常用的方法是构建单细胞悬液。
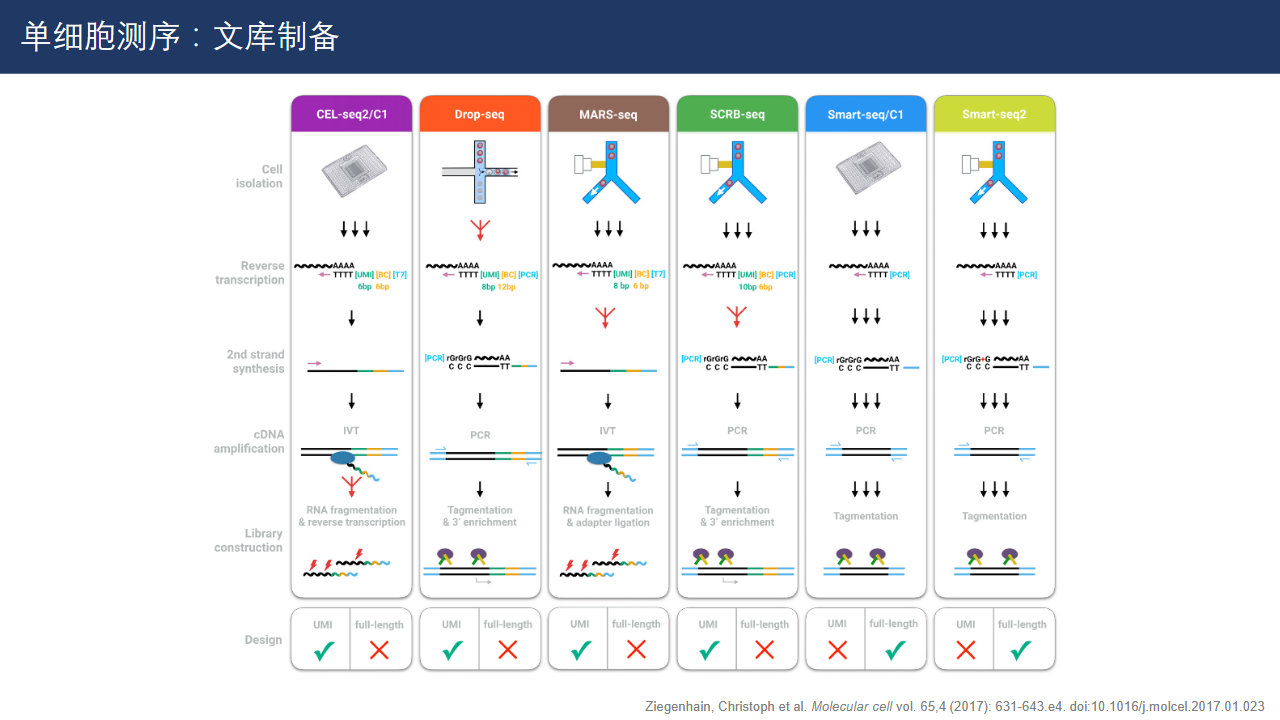
针对不同需求,有不同的文库制备方法。其中最常用的包括10x genomics Drop-seq(油包水构建单个细胞的扩增反应体系)、Smart-seq(微孔测序)。
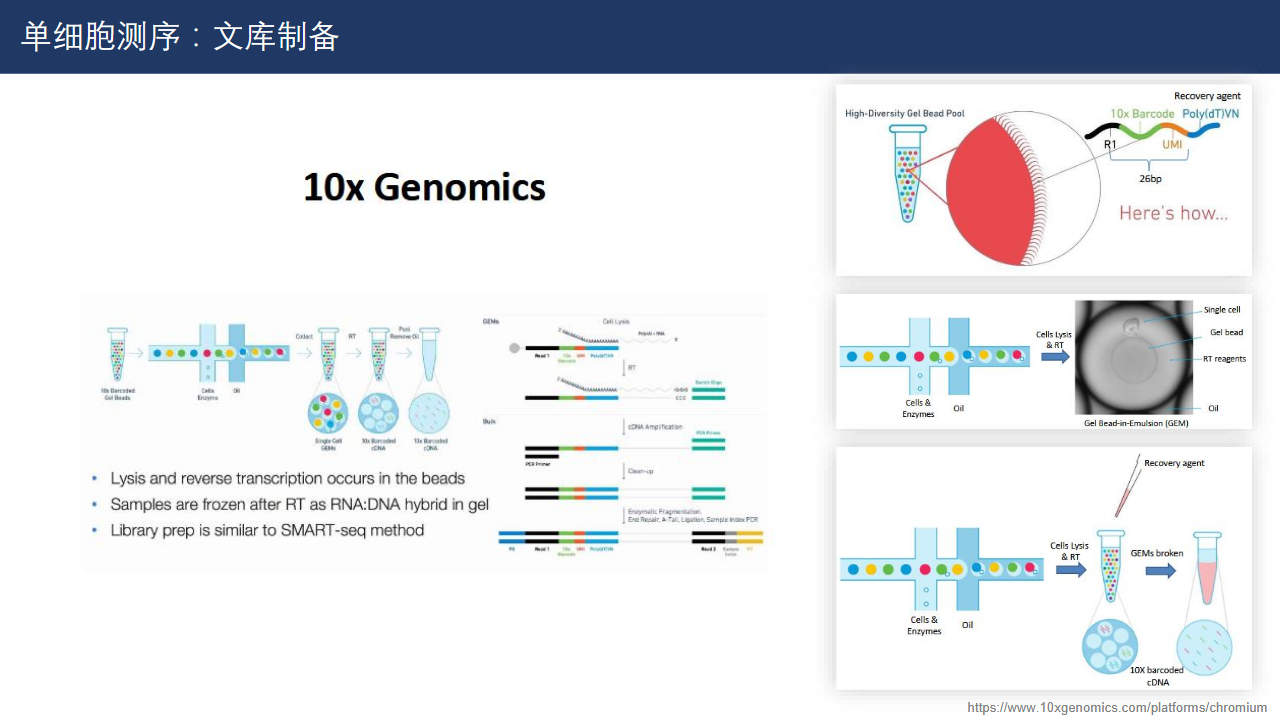
上图展示了10x genomics drop-seq构建油包水反应体系的原理。
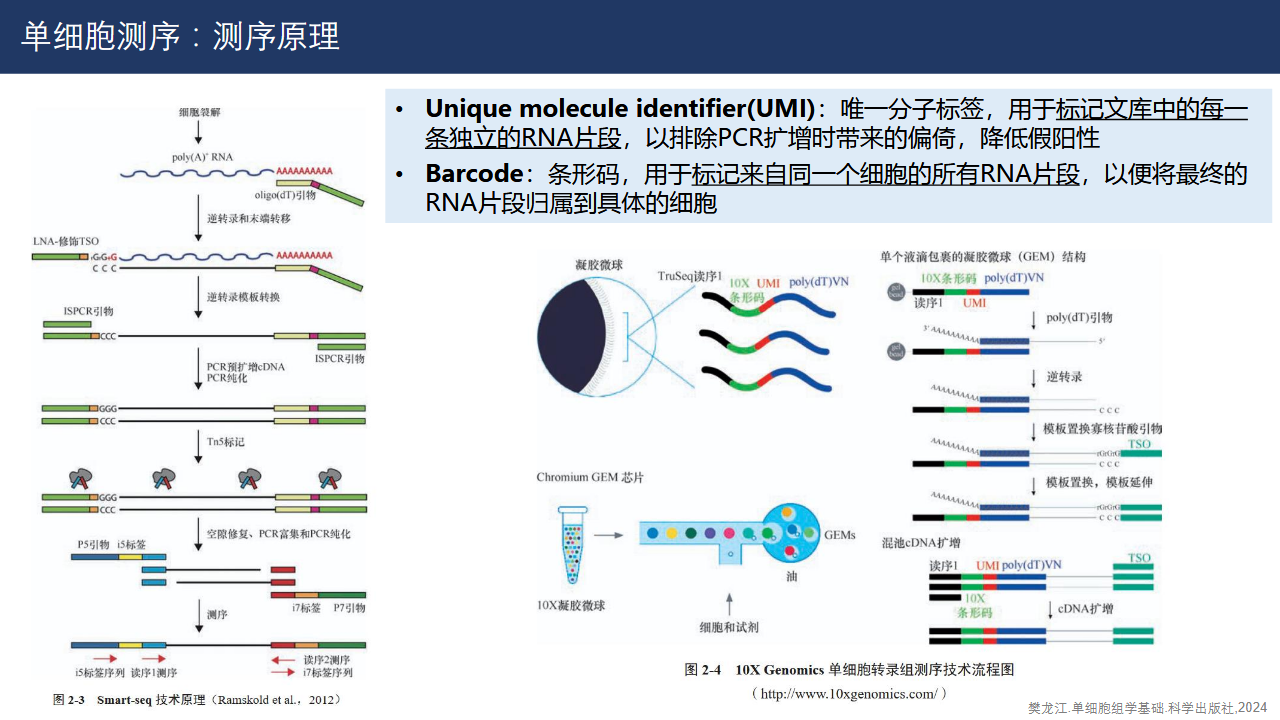
区分两个重要概念:UMI vs Barcode
- Unique molecule identifier(UMI):唯一分子标签,用于标记文库中的每一条独立的RNA片段,以排除PCR扩增时带来的偏倚,降低假阳性【也就是说区分每一条独特的reads】
- Barcode:条形码,用于标记来自同一个细胞的所有RNA片段,以便将最终的RNA片段归属到具体的细胞【也就是说区分单个细胞】
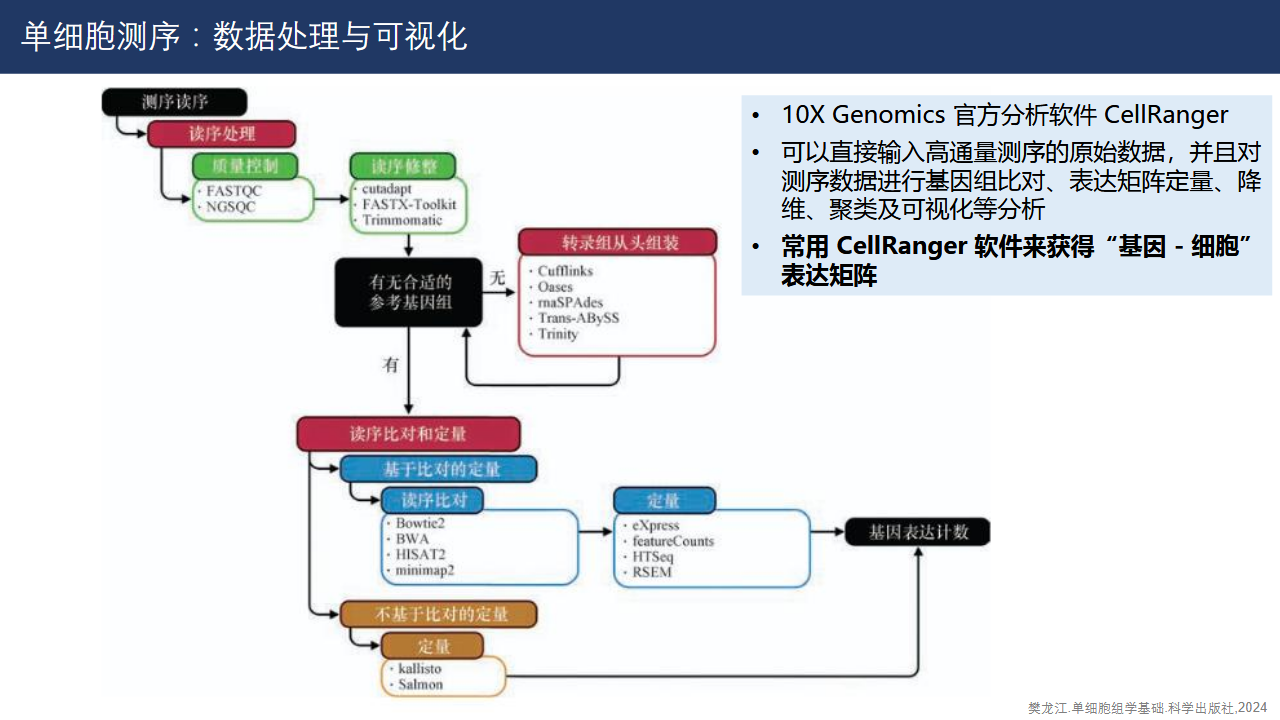
数据处理和可视化的pipeline(流程图)。具体涉及的软件见下文以及后续blog更新。
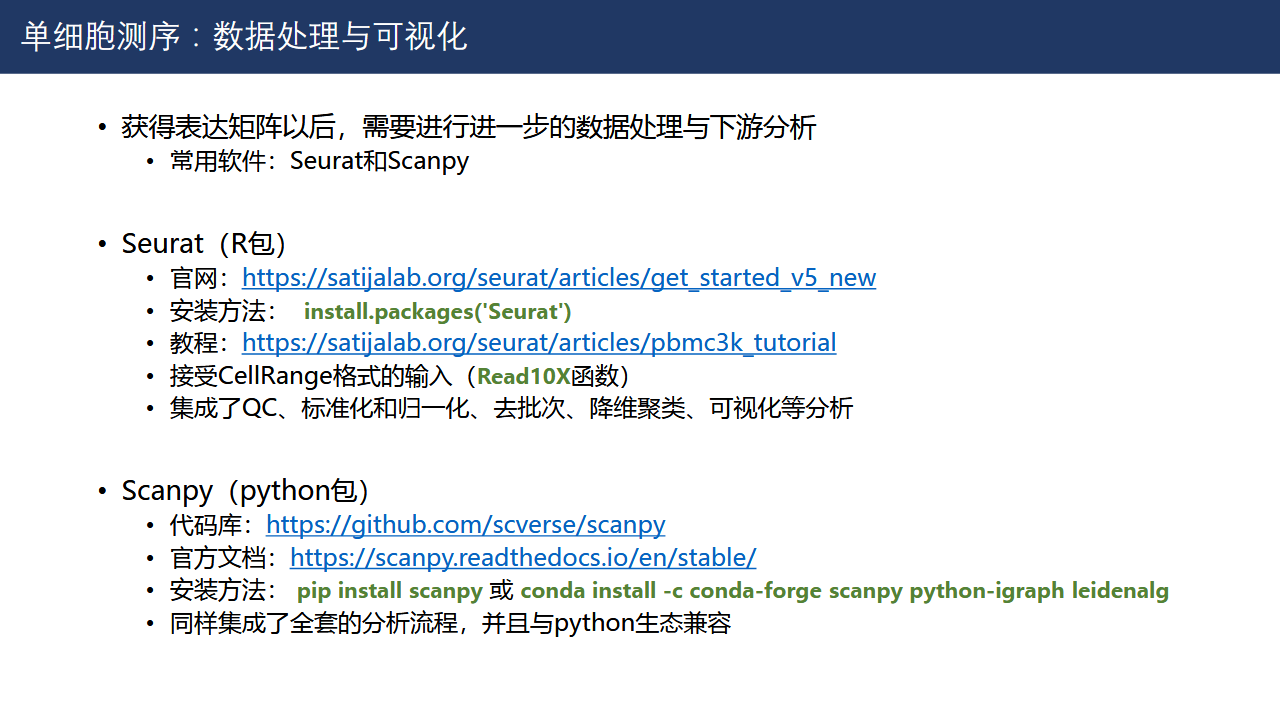
两大主流软件:Seurat vs Scanpy

step-by-step的分析流程

样本整合与批次效应矫正技术。另外参考博客往期文章:《单细胞数据处理中的归一化和去批次效应》
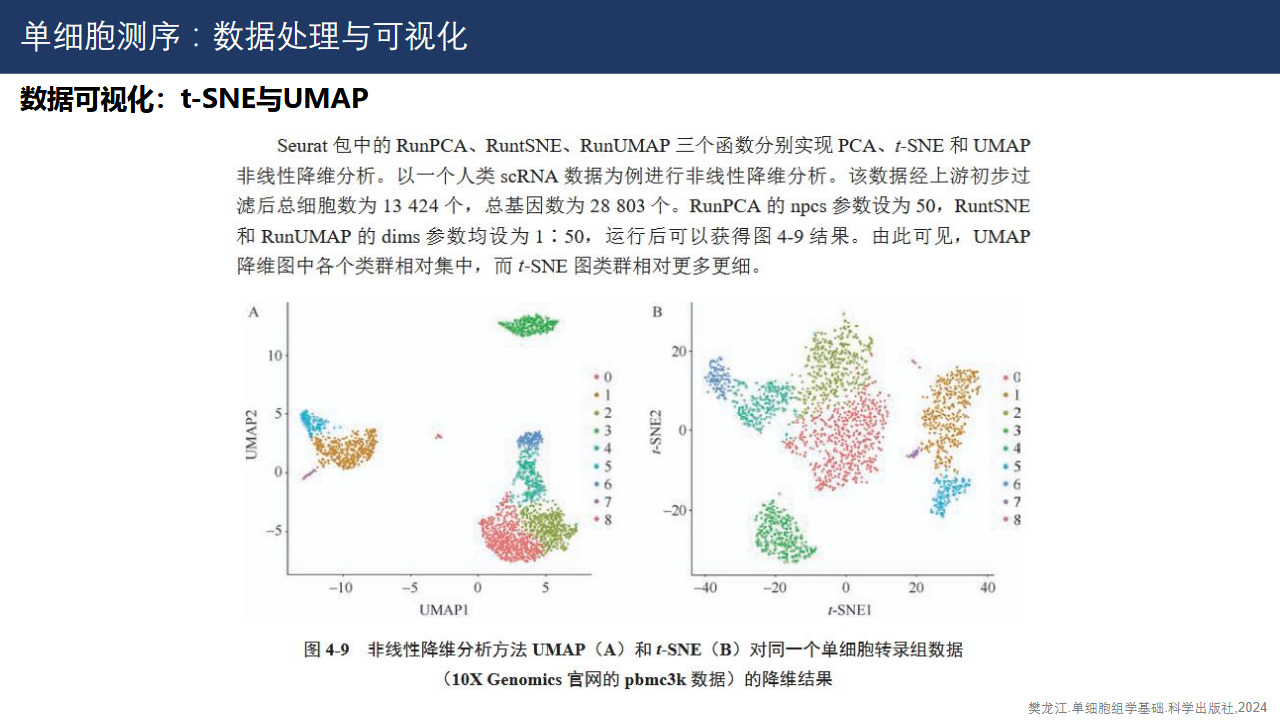
细胞分群可视化:常用t-SNE和UMAP两种降维方法。
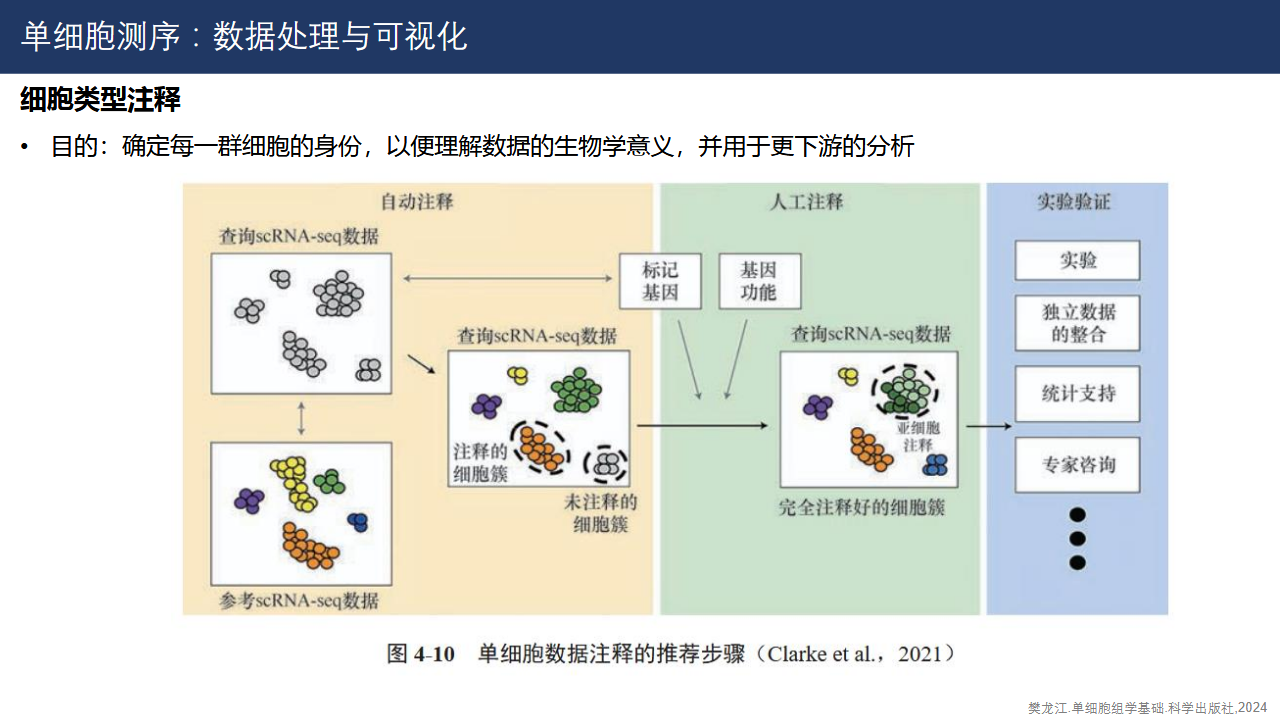
细胞类群注释。这是单细胞分析中的重要步骤之一。
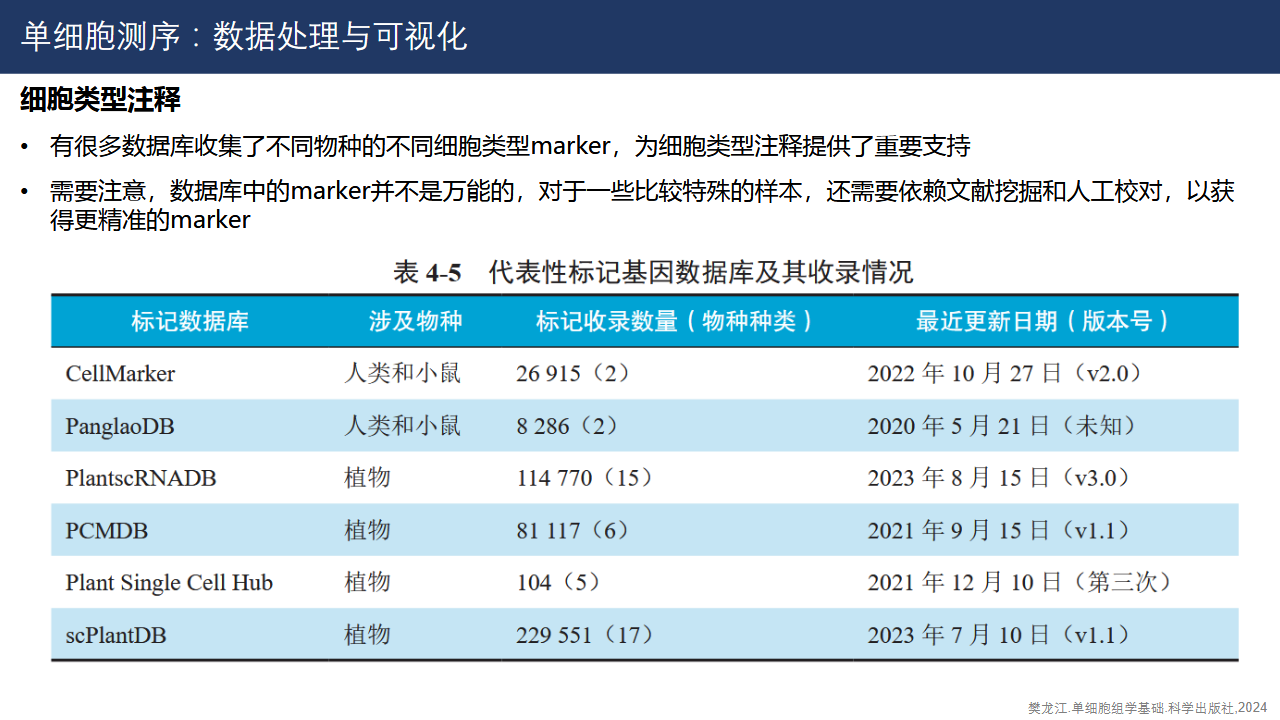
常用注释biomarker数据库。
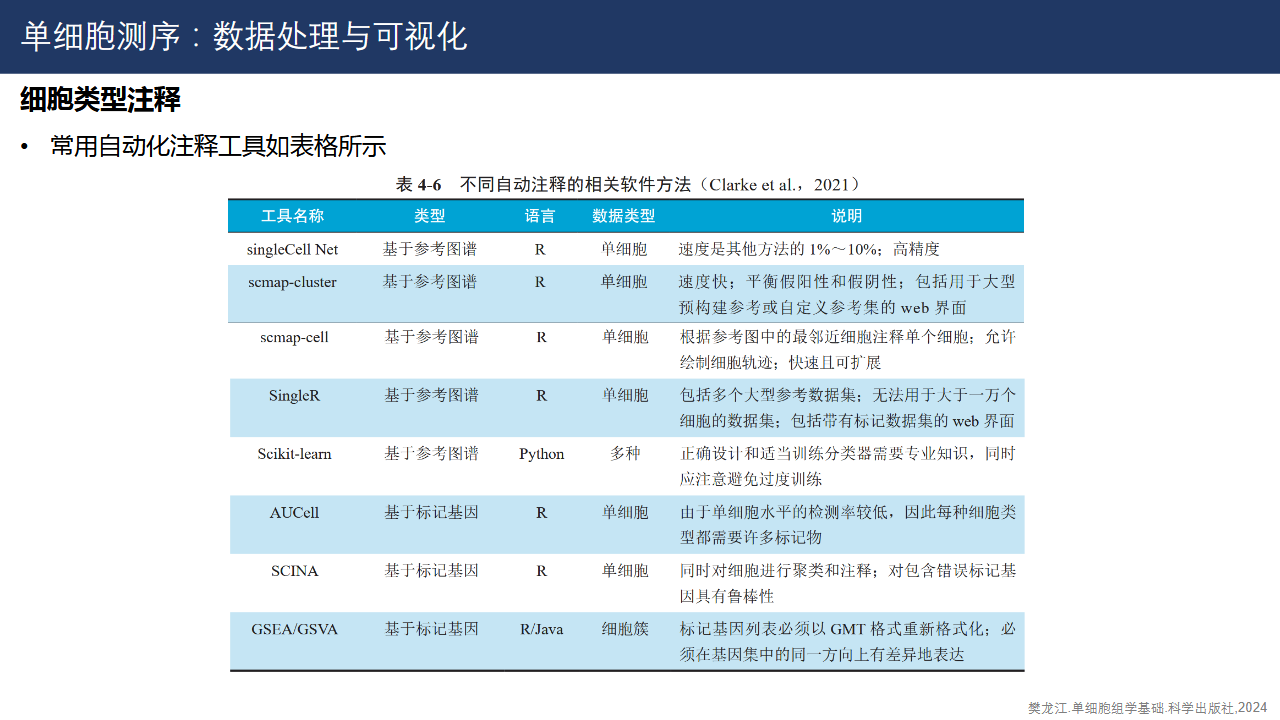
常用自动化注释工具。
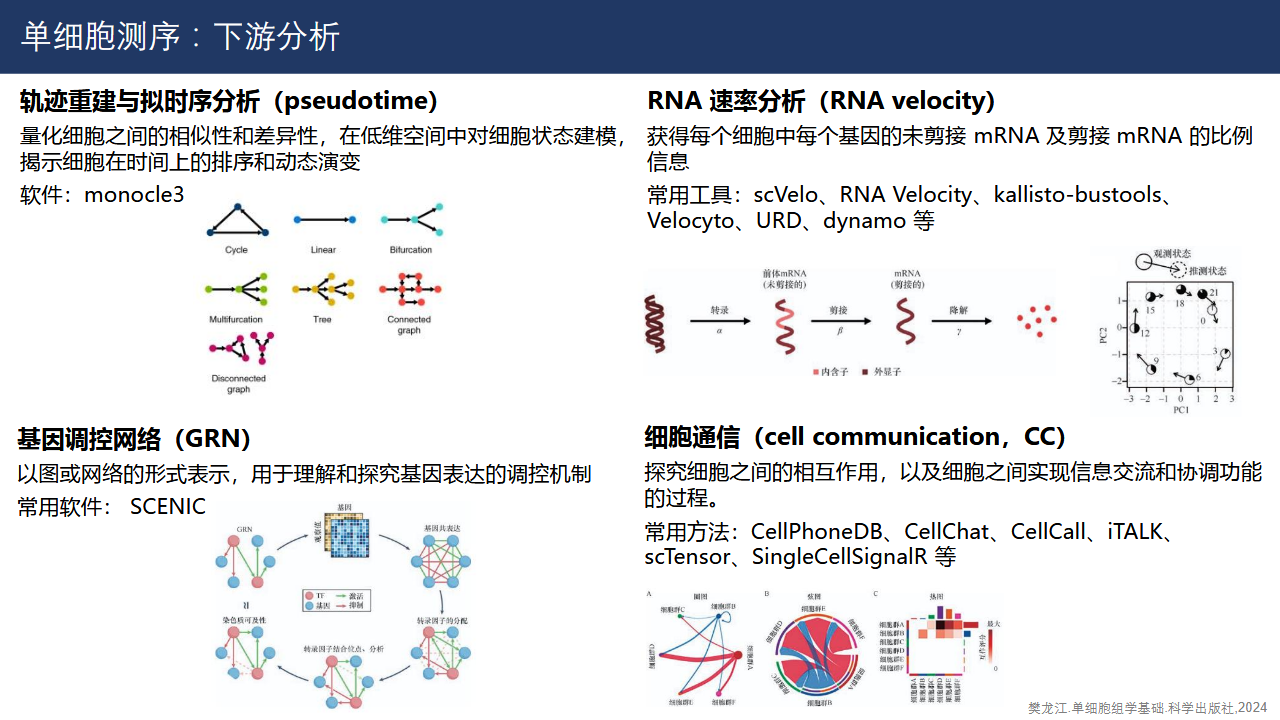
有许多下游分析技术。这些下游分析技术可以帮我们从不同层面理解单细胞层面的动态。

这是老师课上的几道思考题。

还有几种工具的比较。
以上。