蛋白质定量工具DIA-NN在Linux系统上的安装与使用
多组学研究已经成为了目前生物学研究中的重要方向之一。而支撑多组学研究的是多组学测序技术,包括DNA测序、RNA测序、RNA微阵列芯片、蛋白质质谱测序等诸多方法。其中,蛋白质组学,由于涉及到生命活动最重要的承载者即蛋白质本身,就显得尤为重要。
传统的蛋白质测序以Edman降解法为代表,原理是通过酶切反应在蛋白质的N端(或C端)逐个分解氨基酸残基,并对分解下来的氨基酸残基进行鉴定,最终组装为一整条多肽序列。这种方法精度很高,但反应复杂、测序速度慢,无法进行大规模高通量检测。近年来快速发展的高通量蛋白质测序方法是质谱法,将蛋白质(或经过特定蛋白酶预先消化的肽段)通过高压电场带上电荷,再通过磁场偏转,测定肽段碎片的质荷比,并比对数据库中已知肽段的数据,最终拼凑出完整肽段的序列(以及相应的蛋白质丰度)。近年来快速发展起来的深度神经网络(Deep Neuron Network,DNN)更为质谱法测序的拼接和比对工作增添了许多活力。
目前常用的蛋白质定量工具包括Spectronaut 、 MaxQuant 、 FragPipe 、 DIA-NN 等。不同软件有不同的特点和使用场景,例如Spectronaut是商业授权软件,功能齐全但价格昂贵;MaxQuant和DIA-NN为开源软件,前者历史悠久兼容性好,后者则引入了深度神经网络效率更高。
本文将简单介绍DiaNN的原理、安装和运行demo的方法。
一、DiaNN介绍
DIA-NN 是一款用于数据非依赖性采集(Data-Independent Acquisition, DIA)蛋白质组学数据分析的软件套件,由剑桥大学的团队Vadim Demichev等人开发。下面是这个工具最初的论文以及Github页面,在GitHub页面上有这一工具的详细使用手册。
citation: Demichev, V., Messner, C.B., Vernardis, S.I. et al. DIA-NN: neural networks and interference correction enable deep proteome coverage in high throughput. Nat Methods 17, 41–44 (2020). https://doi.org/10.1038/s41592-019-0638-x
github: vdemichev/DiaNN
(一)什么是DIA
DIA(Data-Independent Acquisition,数据非依赖性采集)是一种质谱仪的操作模式或技术流派,而不是一种数据格式或单纯的数据处理方法。它与另一种主流技术DDA(Data-Dependent Acquisition,数据依赖性采集)相对。
- 在传统的 DDA 模式下,质谱仪会先进行一次扫描(MS1),检测所有离子的信号强度。然后,它会“智能地”选择信号最强的前N个离子(通常是肽段),将它们隔离并打碎(碎片化),再对这些碎片离子进行第二次扫描(MS2),生成一张针对这N个特定母离子的“峰图”(Spectrum)。这个过程是“依赖性”的——它依赖于上一轮扫描中哪些离子信号强。
- 而在 DIA 模式下,质谱仪会放弃这种“选择强者”的策略。它会将整个质量范围划分成一系列连续的小窗口(例如,每次覆盖4 Da的质量范围),然后循环地、不加选择地对每一个窗口内的所有离子同时进行隔离和碎片化,并记录下所有产生的碎片离子信息。这样做的结果是,每一次循环都会产生一个复杂的混合碎片谱图,其中包含了该质量窗口内所有共洗脱肽段的碎片信息。
(二) DIA-NN的原理
DIA-NN 的核心是利用深度神经网络(Deep Neural Networks, DNNs)来区分真实的质谱信号与背景噪声,并结合新的定量和信号校正策略。
- 分析流程:DIA-NN 的工作流程始于一个以肽段为中心的方法。它可以从一个已有的光谱库中获取信息,或者直接从蛋白质序列数据库(FASTA文件)中自动进行_in silico_(计算机模拟)生成一个预测的光谱库(即“无库模式”)。
- 信号识别与去噪:软件会为每个目标或“诱饵”(decoy,用于计算假阳性率FDR)前体离子提取色谱图,并在预估的洗脱峰附近寻找由前体离子和碎片离子共同构成的信号。DIA-NN 使用的深度神经网络能更有效地从复杂的背景噪音中识别出真实的肽段信号。
- 干扰校正:DIA-NN 采用了一种独特的策略,可以有效处理共洗脱肽段之间的碎片离子干扰问题。它会评估不同前体离子间的碎片干扰程度,并报告其中得分最高的那个,从而结合了“肽段中心”和“谱图中心”两种分析方法的优点。
- 定量与归一化:DIA-NN 实现了名为 QuantUMS 的机器学习优化定量模式,旨在最大化定量精度,同时减少比值压缩(ratio compression)现象。它还会对技术变异(如上样量差异)进行归一化处理,以突出生物学差异。
- 统计控制:通过创建“诱饵”库并严格计算q值(false discovery rate, FDR),DIA-NN 能够对肽段和蛋白质的鉴定提供严格的统计学置信度评估。
需要注意的是,虽然DIA-NN原生为分析DIA数据而开发,但在最新版本(2.3)中已经引入了对DDA测序模式的数据的支持,只需要在命令行中添加 --dda 参数即可。
(二)在Linux中以docker方式安装DIA-NN
这一部分的工作笔者花了很多时间探索。DiaNN基于微软的 .NET framework 技术开发,原生运行在Windows系统上,且有一个优秀的GUI界面(如下图);但是我们的数据在Linux服务器上,下载下来不现实。如何在Linux服务器上运行DIA-NN成为了一个问题。
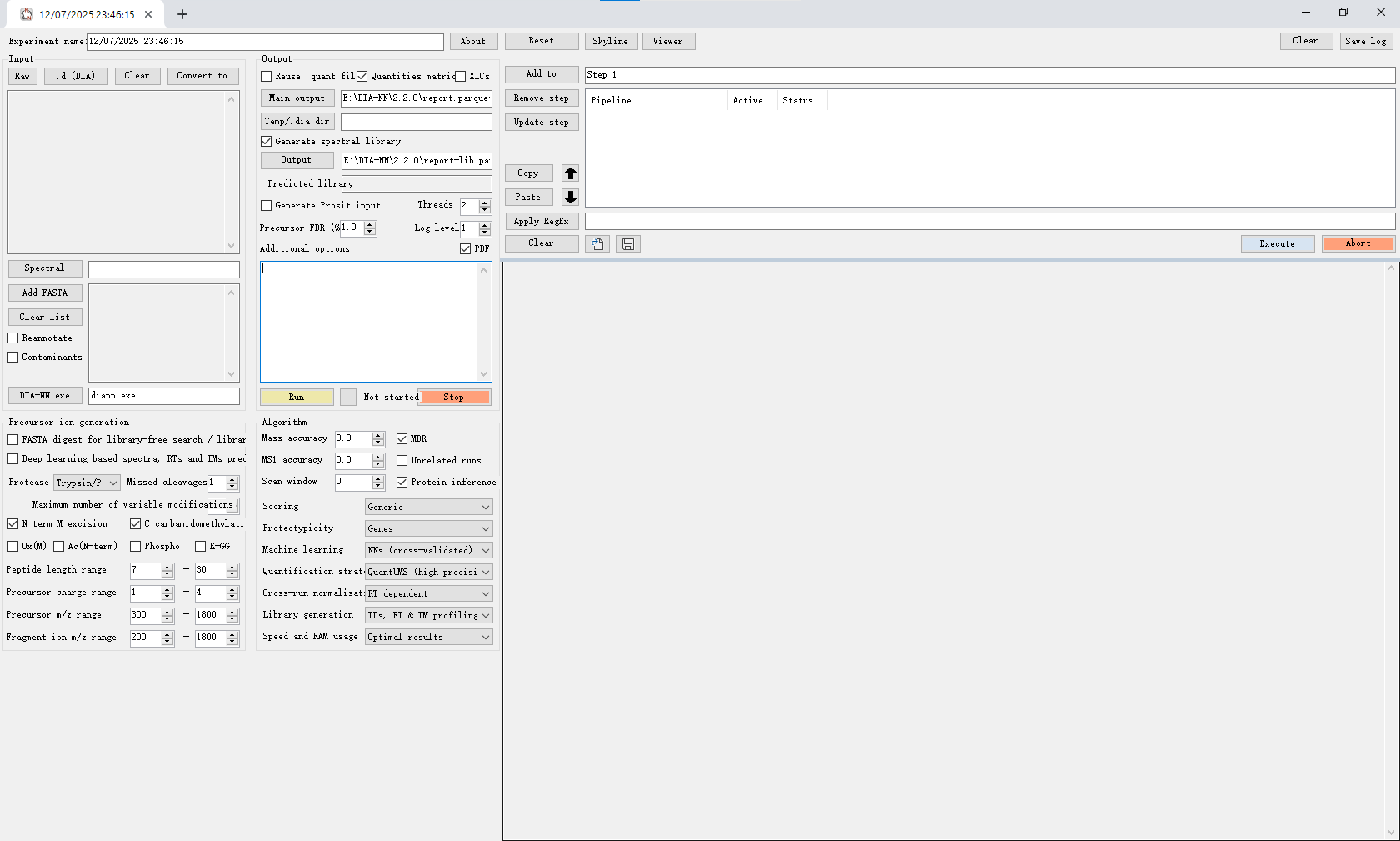
官方文档中给出了在Linux中安装的方法,如下段落所示。其需要在Linux系统中预先安装.NET SDK 8——对于实验室的服务器集群来说没有root权限很难实现这一点。另一种退而求其次的方法是通过docker运行,在DIA-NN的安装包中提供了docker的脚本,可以进行安装。
On Linux (command-line use only), download and unpack the Linux .zip file. The Linux version of DIA-NN is generated on Linux Mint 21.2, and the target system must have the standard libraries that are at least as recent plus .NET SDK 8.0-series, version 8.0.407 or later. There is no such requirement, however, if you make a Docker or Apptainer/Singularity container image. To generate either container, we recommend starting with the latest debian docker image, the script make_docker.sh that does this is included. You can also check the excellent guide by Roger Olivella. See Command interface for Linux-specific usage guidance.
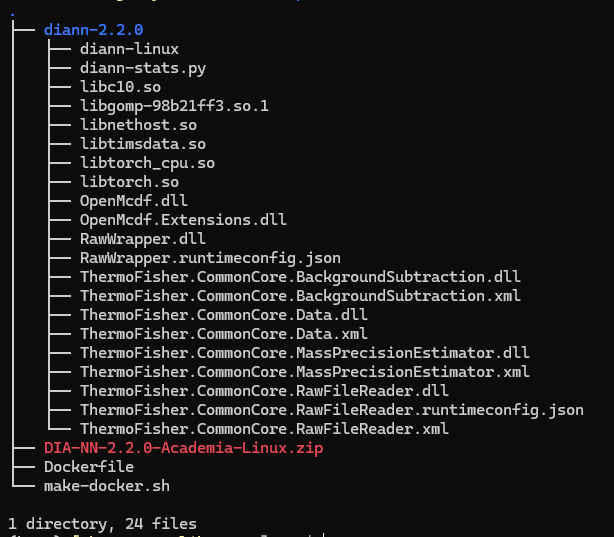
下图为DIA-NN的Linux安装包的内容。其中, diann-2.2.0/diann-linux 是主程序(但需要依赖.NET SDK 8,直接运行是运行不了的);Dockerfile和 make-docker.sh 是创建docker镜像所需的配置文件。
其中,make-docker.sh 文件只有一行(如下所示),看起来是调用docker指令创建了一个镜像容器,容器的名称为 diann_docker 即为我们最终要用的容器名称。
1 | docker build --no-cache -t diann_docker . |
Dockerfile的内容如下所示。左边是原始Dockerfile,总共大约分为4个步骤,分别实现了环境配置和软件安装;由于安装过程中网络连接出错,我对Dockerfile进行了修改,最终的Dockerfile如右图。
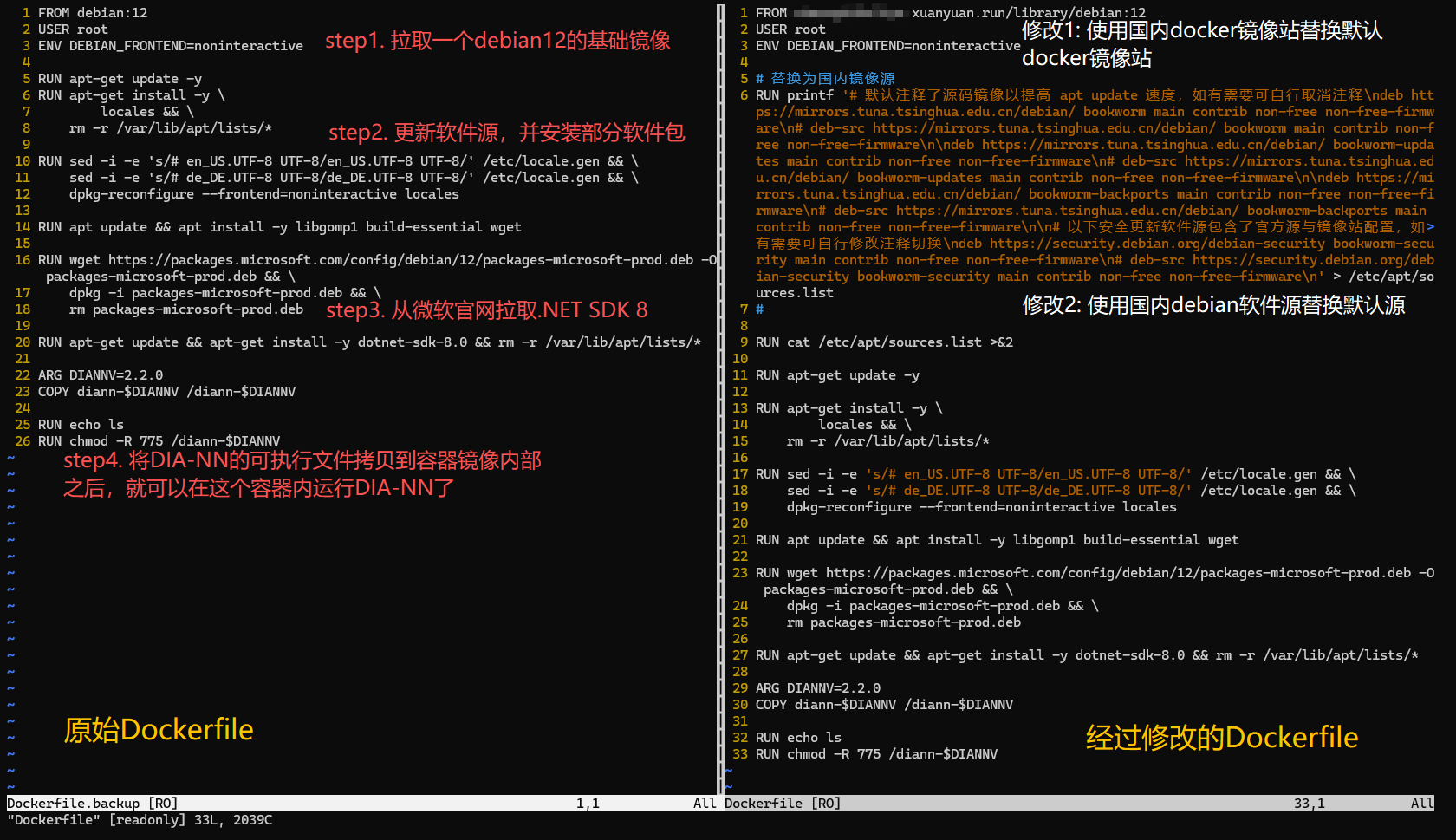
修改过的Dockerfile内容如下:
1 | FROM ckmrogdt0elhyw.xuanyuan.run/library/debian:12 |
这里多说一句:鉴于docker官方镜像站的网络访问问题,我注册了轩辕云(xuanyuan.run)的账户,并使用轩辕云镜像站作为dockerhub地址,因此在拉取镜像的这一步使用的镜像名称为 ckmrogdt0elhyw.xuanyuan.run/library/debian:12 而不是 debian:12。
对于我们用户来说,DIA-NN的安装只需要一步(前提是Dockerfile已经修改好了;否则整个过程中的网络问题会给我们带来许多困扰):
1 | bash ./make-docker.sh |
安装过程大约需要几分钟,最终完成以后会输出下面这样的安装日志:
1 | => [11/12] RUN echo ls 0.3s |
还可以使用下面的指令测试是否安装成功:
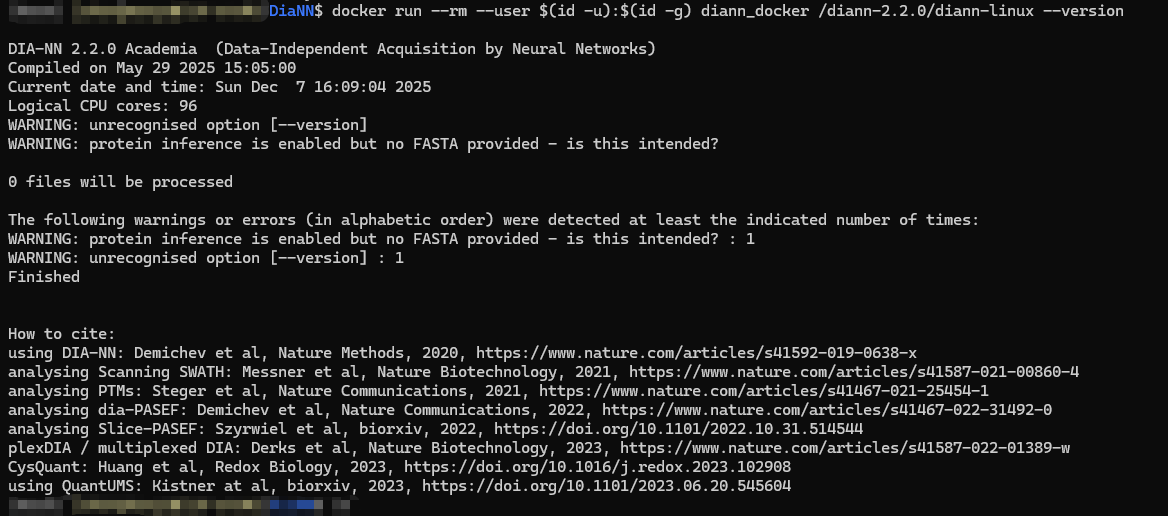
1 | docker run --rm --user $(id -u):$(id -g) diann_docker /diann-2.2.0/diann-linux --version |
如果安装成功,则上述指令会产生如下的输出:
三、在Linux上使用DIA-NN:一个简单的demo
前面我们提到,如果想要查看DIA-NN是否安装成功(或者仅仅是查看版本号),可以运行下面的指令:
1 | docker run --rm --user $(id -u):$(id -g) diann_docker /diann-2.2.0/diann-linux --version |
而在使用DIA-NN分析数据的时候,我们只需要把 --version 这一部分替换为实际分析的指令参数即可。
下面是一个具体的例子(写在 run_diann_quantification.sh 文件当中)。我们需要准备输入文件(.d格式的文件夹)和fasta库文件。fasta库文件(参考蛋白组文件)是必需的,这个fasta库文件可以从uniprot下载,注意需要与样本的物种对应。
1 |
|
另一个需要注意的地方是,许多实验室的高性能计算集群服务器使用了网络文件系统(Net File system,NFS)以方便存储空间拓展以及服务器托管,但是docker并不能很好的处理跨NFS或者跨软链接的文件访问,因此读者在运行前需要先确认一下文件路径中是否存在跨NFS的路径或者跨软链接的路径,如果存在,需要统一一下路径,或者以系统 /tmp 目录作为临时中转站。
在分析流程结束后,DIANN会在result目录下生成多个文件,包含了分析过程的一些中间文件以及最终的定量结果。在这些文件当中,最重要的文件有三个:
| 文件名 | 作用 |
|---|---|
| *.pg_matrix.tsv | 这是核心文件,包含表达矩阵信息,后续做差异分析就靠它 |
| *.pr_matrix.tsv | 肽段层面的矩阵,质控用 |
| *.parquet | 包含所有细节以及中间过程的大表,以备不时之需 |
以上。
最后,祝大家代码全跑通,可以准时下班awa。