《自然》论文:多个重叠的结合位点决定转录因子占据情况
这周组会上讲述的一篇论文。作者提出了一种检测蛋白质和DNA之间低亲和力相互作用的技术PADIT-seq,并通过这种技术发现了转录因子调控中的两种新机制。
Citation:Khetan, S., Carroll, B.S. & Bulyk, M.L. Multiple overlapping binding sites determine transcription factor occupancy. Nature (2025). doi:10.1038/s41586-025-09472-3
本文另见微信公众号报道:
Nature | DNA的“俄罗斯套娃”:PADIT-seq揭示了一个颠覆教科书的转录因子结合新模型
背景
(一)作者信息:
本文的作者团队均来自哈佛大学医学院(更详细的信息可见 课题组官网 )。研究团队聚焦于转录因子结合的生物学机制研究,开发了大量技术和软件工具,自2002年起,已发表近百篇论文和十多种软件工具。

(二)前人研究:检测蛋白质-DNA互作的技术
文章中提到了如下技术:
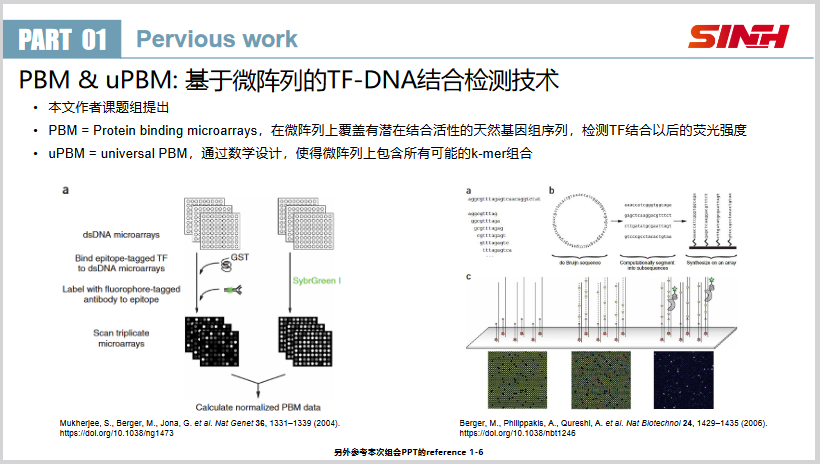
- PBM:一种基于微阵列的检测技术,可以大规模检测一种蛋白质和不同DNA的互作。 优点 :高通量;成本低(并且为本文实验室首创,有大量技术积累)。 缺点 :只能检测高亲和力的DNA片段,对于低亲和力片段的敏感性不足。
- uPBM:是PBM的变种方法。PBM的微阵列上附着的是天然存在的DNA片段,但是uPBM上附着的是经过数学计算的所有可能的DNA序列组合。 因此理论上uPBM可以检测一种特定转录因子的所有潜在DNA结合片段。
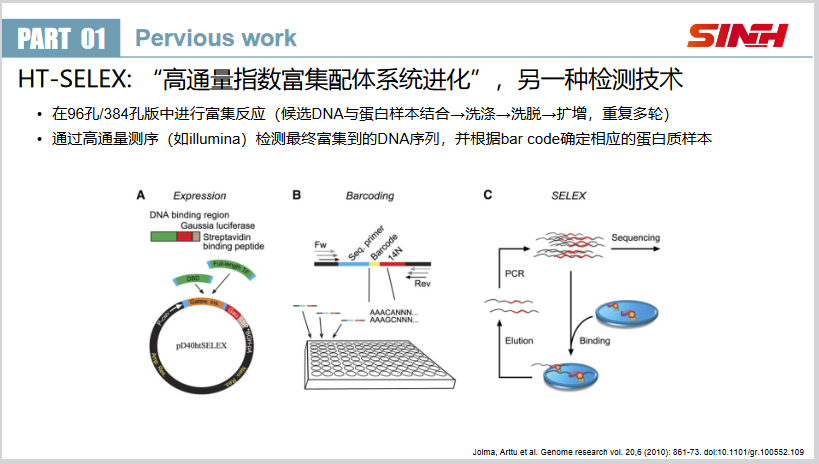
- HT-SELEX:基于多轮扩增反应,对高亲和力DNA片段进行富集。优缺点和PBM相同。
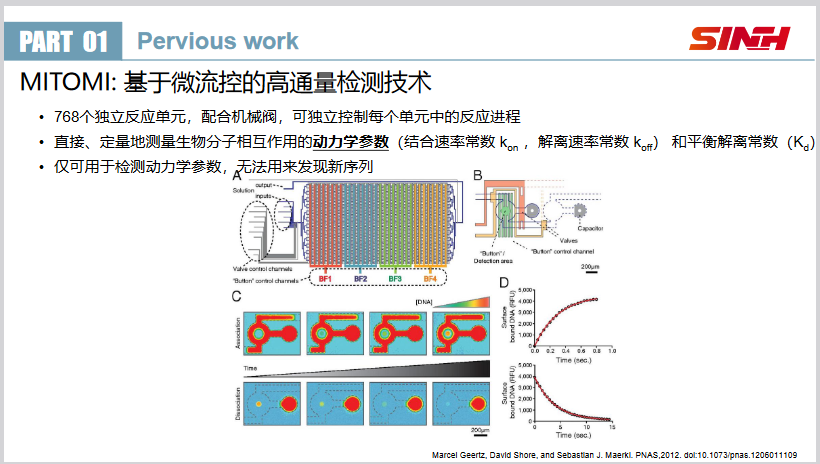
- MITOMI:一种基于微流控系统的检测技术。优点 :可以精确定量蛋白质和DNA之间的作用力强度(以化学平衡常数 $K_d$ 计)。 缺点 :测序通量较低,并且无法用于发现未知的高亲和力片段。
- BET-seq:一种基于微流控系统的高通量检测技术,相比于MITOMI改进了控制系统和测序方法,并在下游分析技术中引入了深度学习模型。优点 :可以精确定量蛋白质和DNA之间的作用力强度(以相对自由能变化 $\text{ΔΔ}G$ 计),测序通量较高,可用于发现未知的高亲和力片段。 缺点 :成本较高。
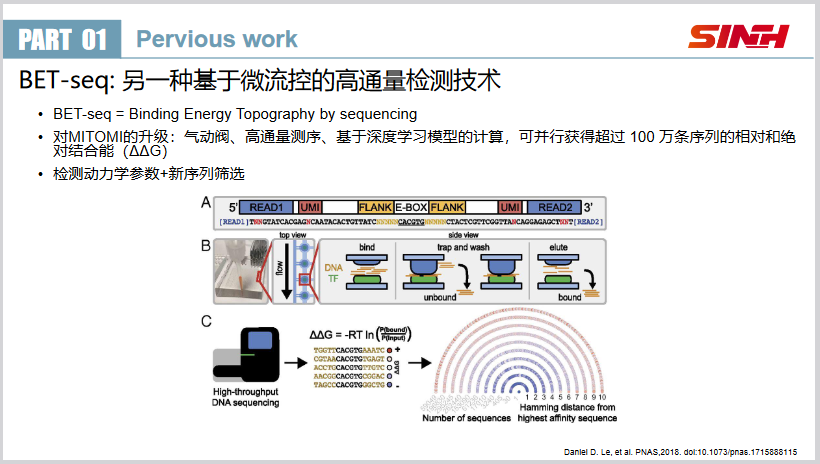
本文的出发点在于,如何寻找那些与转录因子有较低亲和力的DNA片段(作者认为这些低亲和力片段也有重要的调控功能),同时降低实验成本。最终,作者提出了PADIT-seq,在体外系统中通过两阶段的转录,实现低亲和力片段的发现。
本文研究结果
(一)提出了一种新方法:PADIT-seq
PADIT-seq = protein affinity to DNA by in vitro transcription and RNA sequencing
这是一种 体外 的TF结合位点筛选方法。(注意和ChIP-seq之类的方法的区别——后者是体内方法)。
虽然先前已经有许多种TF-DNA binding检测方法,但作者提出的新方法旨在克服现有技术的以下局限:
- 灵敏度不足 (Low Sensitivity):
- 如PBM和HT-SELEX等主流高通量方法,由于其检测原理(荧光信号强度或富集倍数),对低亲和力的结合事件不敏感。它们的检测阈值较高,导致大量真实的低亲和力位点被当作“噪音”而忽略。
- 功能相关性不明确 (Lack of Functional Relevance):
- PBM测量的是“结合”,MITOMI/BET-seq测量的是“亲和力/能量”,HT-SELEX测量的是“富集”。但这些物理化学上的测量结果是否直接对应于功能性的相互作用(即能驱动下游生物学过程)并不总是清晰的。一个微弱的结合可能没有实际功能意义。
PADIT-seq的原理如下图所示:
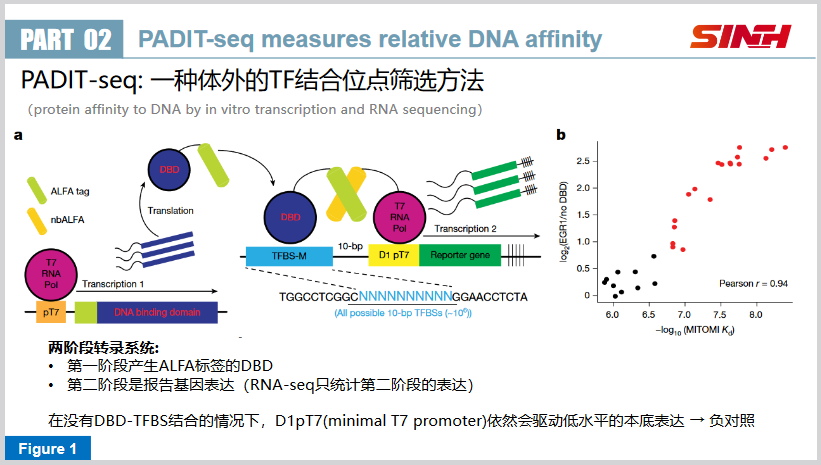
- 第一阶段的转录产生一个带有ALFA标签的转录因子融合蛋白(DBD就是转录因子的DNA结合结构域),这些融合蛋白将对第二阶段的转录产生影响。
- 第二阶段的转录由D1pT7启动子调控,这是一个人为修改的T7启动子:
- 在没有转录因子的条件下,D1pT7会启动低水平转录,产生本底水平的报告基因表达
- 当融合蛋白(DBD)与候选的DNA片段(TFBS-M)发生结合时,融合蛋白的ALFA标签就会招募T7-RNA聚合酶,启动更高水平的报告基因表达
- 报告基因的表达水平与DBD-TFBS结合的亲和力成正比——因此可以检测低水平的DNA亲和力片段。
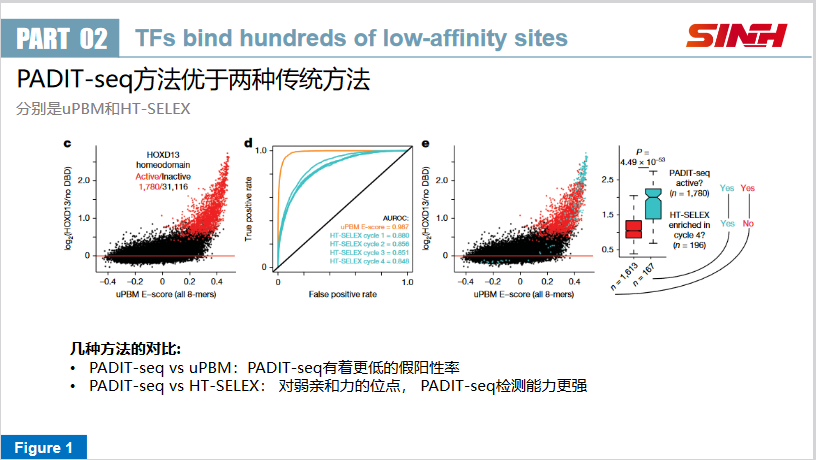
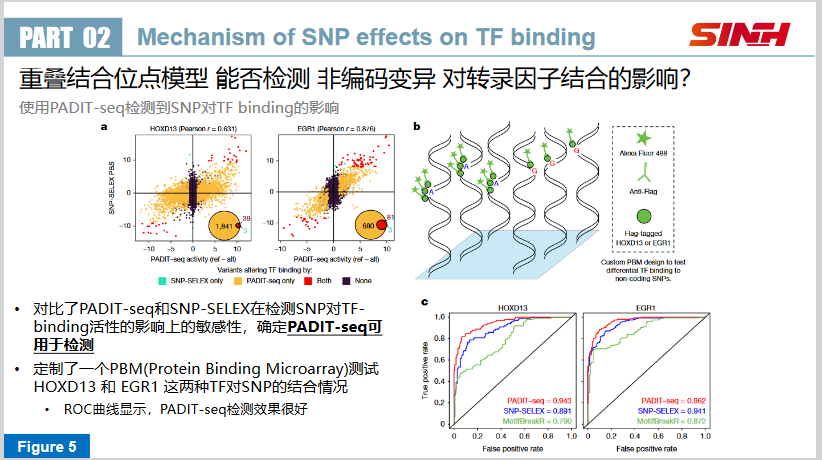
作者对比了PADIT-seq和另外两种传统方法(uPBM和HT-SELEX)。如Fig1c所示,PADIT-seq在假阳性率的控制上优于uPBM;如Fig1d-e所示,PADIT-seq能够找到比HT-SELEX更多的低亲和力位点。
作者还发现,这些低亲和力位点,并不会决定转录因子是否结合,但是能够影响结合的水平。
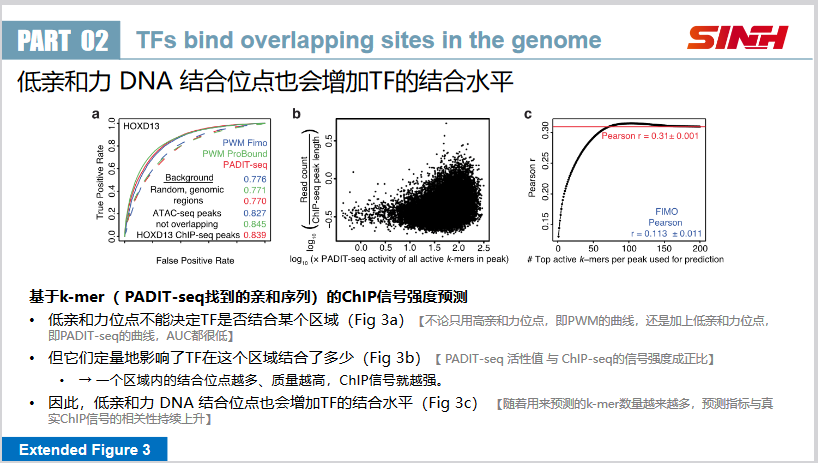
(二)转录因子在基因组中结合重叠的位点
作者使用PADIT-seq,以1bp的步长,对ChIP-seq的peak区域的DNA序列进行滑窗检测。发现在高亲和力位点两侧存在重叠的低亲和力位点。
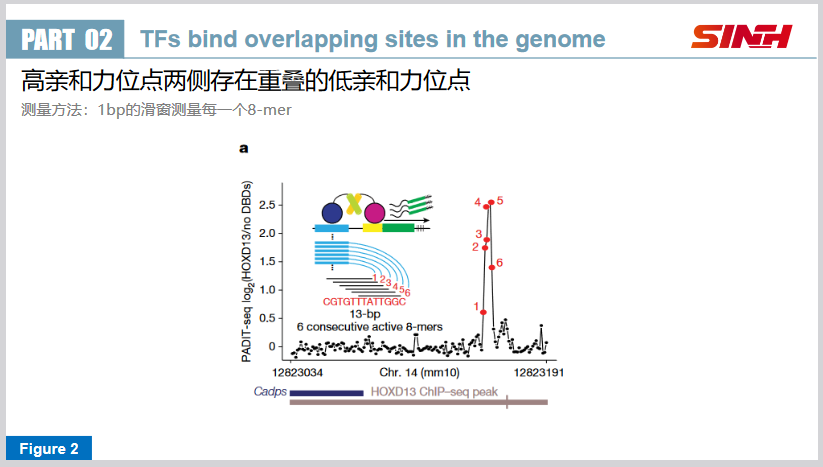
另外,如Fig2b所示,相比于背景基因组,ChIP-seq的peak区域,倾向于富集更多的连续亲和位点,这表明连续位点的存在可能对于转录因子的结合是重要的。
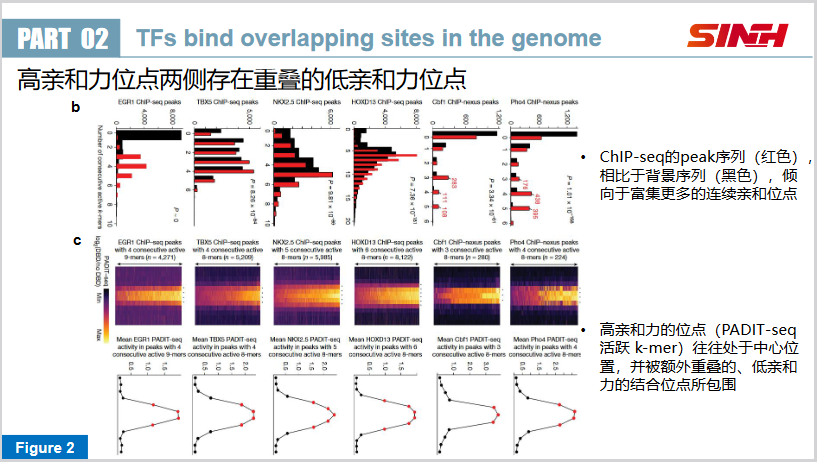
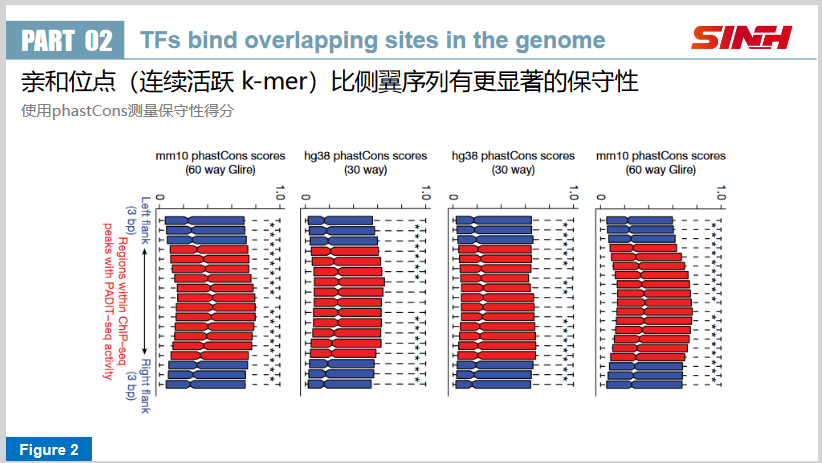
并且这些位点的保守性也更强。
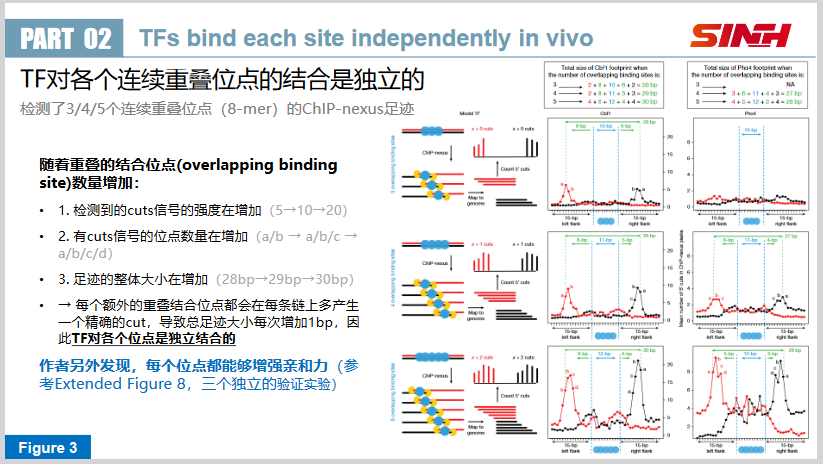
(三)转录因子对于每个重叠位点的结合是互相独立的
如下图。ChIP-nexus是一种能够以1bp分辨率检测DNA上的转录因子结合区域边界的技术。每个“cut”的位置代表TF与DNA的一个精确的接触边界。
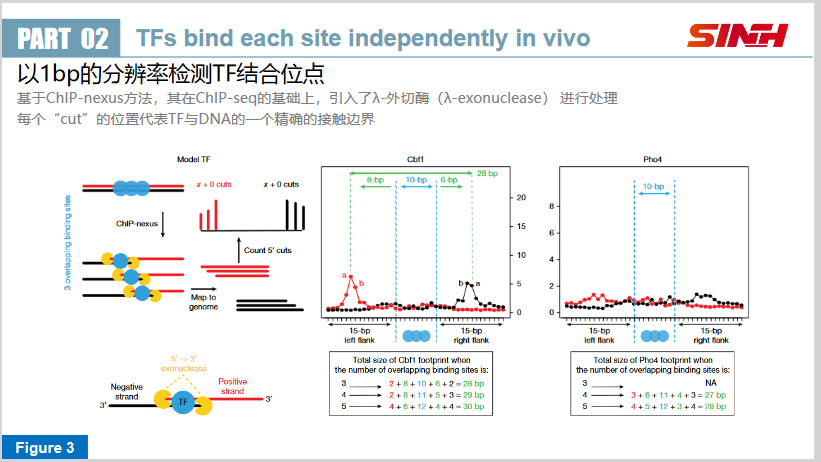
作者发现,随着重叠的结合位点(overlapping binding site)数量增加:
- 检测到的cuts信号的强度在增加(5→10→20)
- 有cuts信号的位点数量在增加(a/b → a/b/c → a/b/c/d)
- 足迹的整体大小在增加(28bp→29bp→30bp)
也就是说,每个额外的重叠结合位点都会在每条链上多产生一个精确的cut,导致总足迹大小每次增加1bp,因此TF对各个位点是独立结合的。
(四)同源转录因子的竞争性结合机制
这是本文的重要生物学发现之一。
同源转录因子意味着它们有着相同的识别基序(例如下图中的Cbf1和Pho4,都识别CACGTG框),那么在实际的转录调控中,它们是如何识别各自特异的结合位点呢?
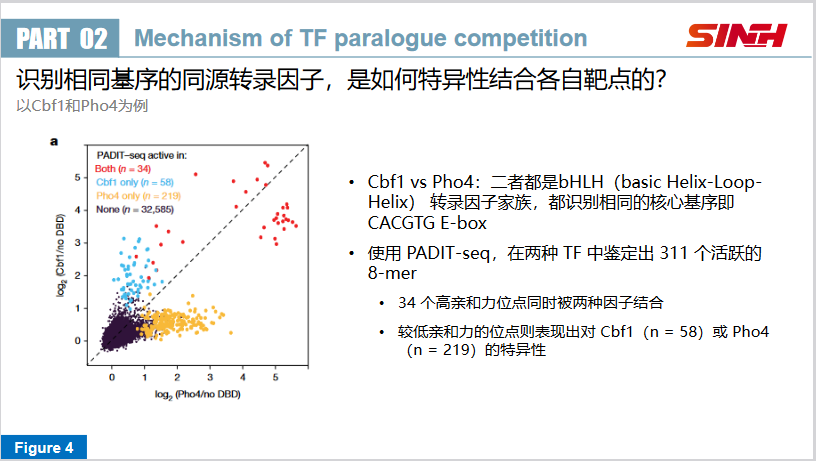
作者首先通过PADIT-seq鉴定出了311个有亲和力的8-mer结合位点序列。其中,有58个是Cbf1特异的,有219个是Pho4特异的。
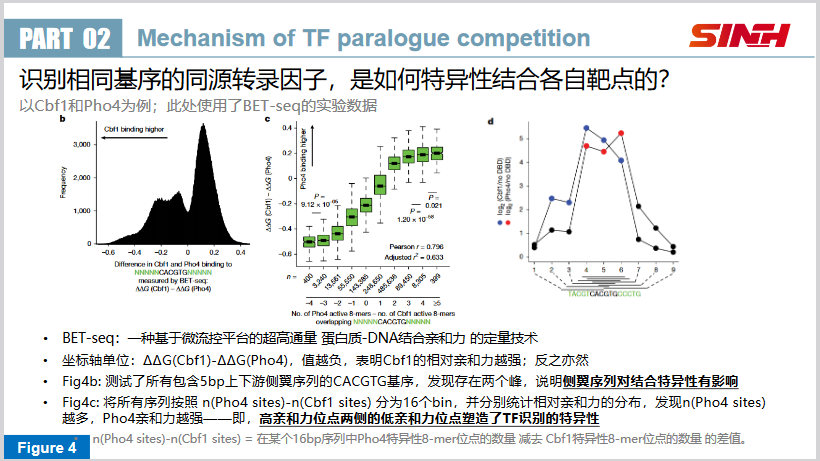
作者使用BET-seq检测了每一种16bp长度的DNA序列组合(其中包括CACGTG这段核心的motif,而侧翼序列则是所有可能的碱基组合)与这两种转录因子的结合亲和力(以相对自由能ΔΔG计)。
- Fig4b:按照相对亲和力的序列频数统计。存在两个峰,说明侧翼序列对结合特异性有影响。(如果没有影响,那么频数分布应该呈现以0为均值的正态分布)
- Fig4c:侧翼序列中,Cbf1特异的8-mer越多,则DNA片段对Cbf1的亲和力越强;反之亦然。也就是说,高亲和力位点两侧的低亲和力位点塑造了TF识别的特异性
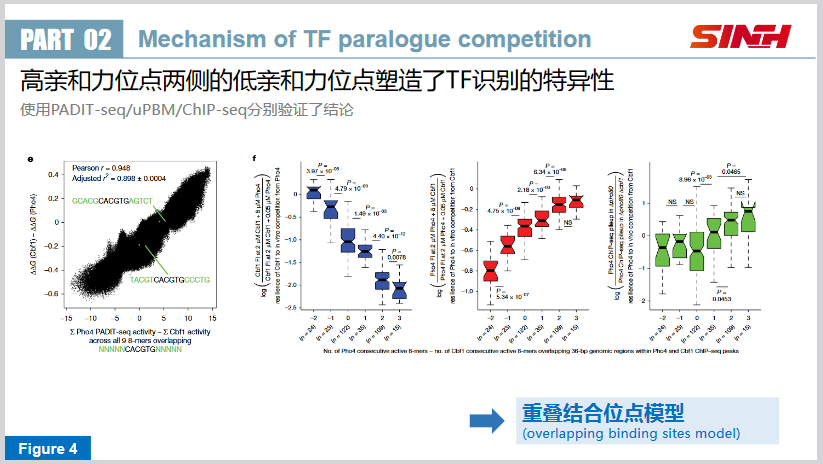
作者还使用另外几种方法分别验证了这一点。最终,作者将这一个机制总结为 重叠结合位点模型(overlapping binding sites model) ,即 高亲和力位点两侧的低亲和力位点塑造了TF识别的特异性 。
(五)突变对转录因子结合的影响
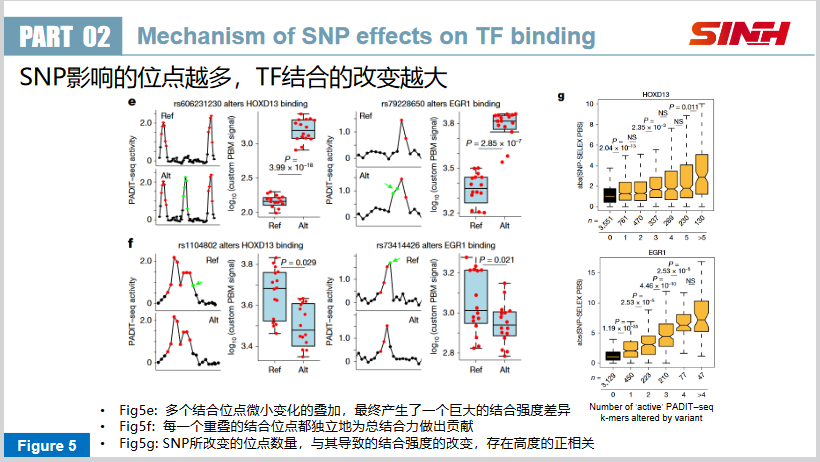
检测了SNP对TF-binding的影响。如下图所示,作者发现,SNP影响的位点越多,TF结合的改变越大。
(六)转录因子结合位点的内在可编织性
所谓“可编织性”(weavable),指的是这些位点之间互相有overlap,可以编织出一张连通图。
如下图,作者提出了一种对TF-binding site进行描述和定量的方法,即网络图——一个节点代表一个k碱基的结合位点(PADIT-seq active k-mers),一条边表示两个节点之间有k-1个bp的序列是重叠的。
作者发现,随机的8-mer序列形成的连通图并不好——最大的单一联通组件只包括7个节点(如下图,左下角)。相比之下,转录因子的连通图,可以形成非常大的单一联通组件(包含80%以上的大部分节点)。
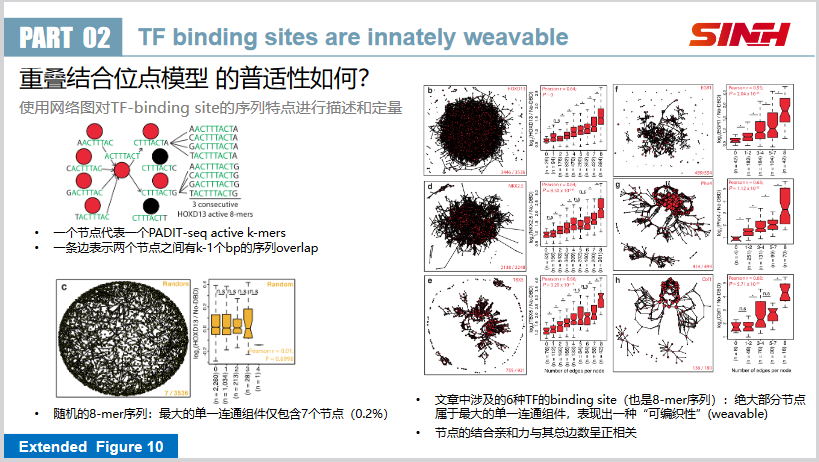
作者还使用 UniProbe 的数据库中的公共数据进行了验证。发现,对于其他转录因子来说,在这样的网络图中也是能够形成极大的单一联通组件(“这些TF 结合位点始终形成了更广泛的网络”)。
这些结果证明:
- “可编织性”是真核生物中不同结构家族的 TFBSs 的固有且普遍的特性
- 重叠结合位点模型很可能代表了真核生物 TF-DNA 相互作用的基本特征。

结论与讨论
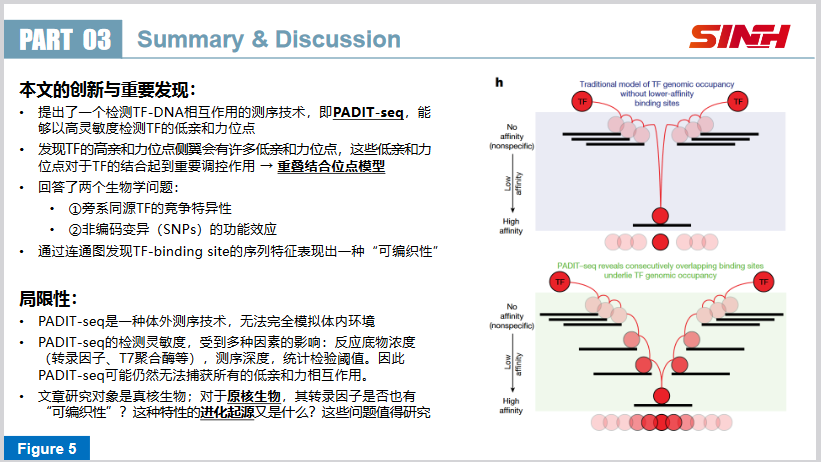
本文的创新与重要发现:
- 提出了一个检测TF-DNA相互作用的测序技术,即PADIT-seq,能够以高灵敏度检测TF的低亲和力位点
- 发现TF的高亲和力位点侧翼会有许多低亲和力位点,这些低亲和力位点对于TF的结合起到重要调控作用 → 重叠结合位点模型
- 回答了两个生物学问题:
- ①旁系同源TF的竞争特异性
- ②非编码变异(SNPs)的功能效应
- 通过连通图发现TF-binding site的序列特征表现出一种“可编织性”
局限性:
- PADIT-seq是一种体外测序技术,无法完全模拟体内环境
- PADIT-seq的检测灵敏度,受到多种因素的影响:反应底物浓度(转录因子、T7聚合酶等),测序深度,统计检验阈值。因此PADIT-seq可能仍然无法捕获所有的低亲和力相互作用。
- 文章研究对象是真核生物;对于原核生物,其转录因子是否也有“可编织性”?这种特性的进化起源又是什么?这些问题值得研究