相关性系数与线性回归
第五次生物统计学助教课的备课笔记。
本文为生统助教课备课过程的一些记录,主要涉及相关性系数、一元线性回归、多元线性回归等知识点。
一张图展示本文知识大纲:
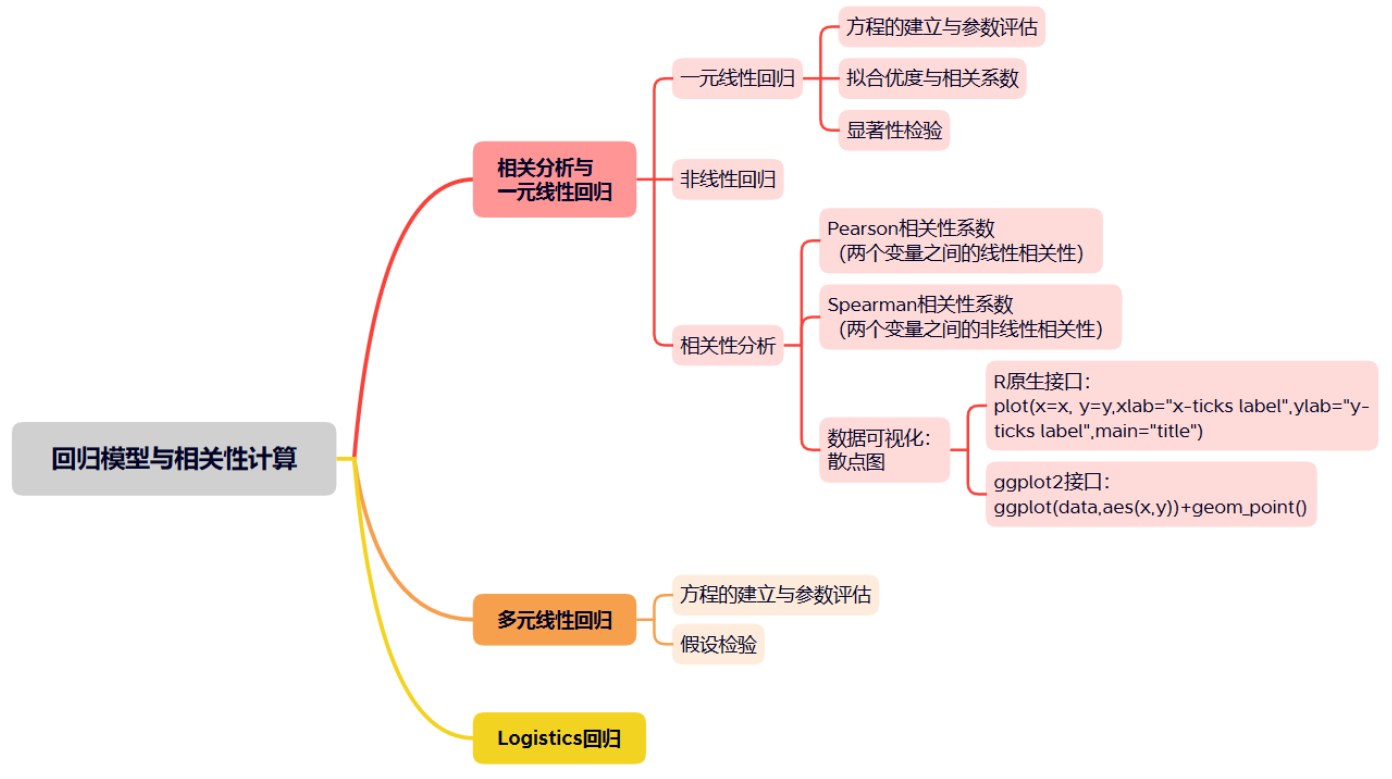
一、一元线性回归与相关性系数
(一)一元线性回归
上一次助教课我们回顾了ANOVA的数学原理,其用来对多组数据的均值差异做检验。线性回归相当于是对ANOVA的进一步拓展,当分组数量是一个连续型变量时,如何检验均值差异的显著性,如何确定自变量(实验因子)对响应变量的影响,就是一元线性回归要解决的问题了。
最小二乘法
使用最小二乘法推导线性回归的公式:

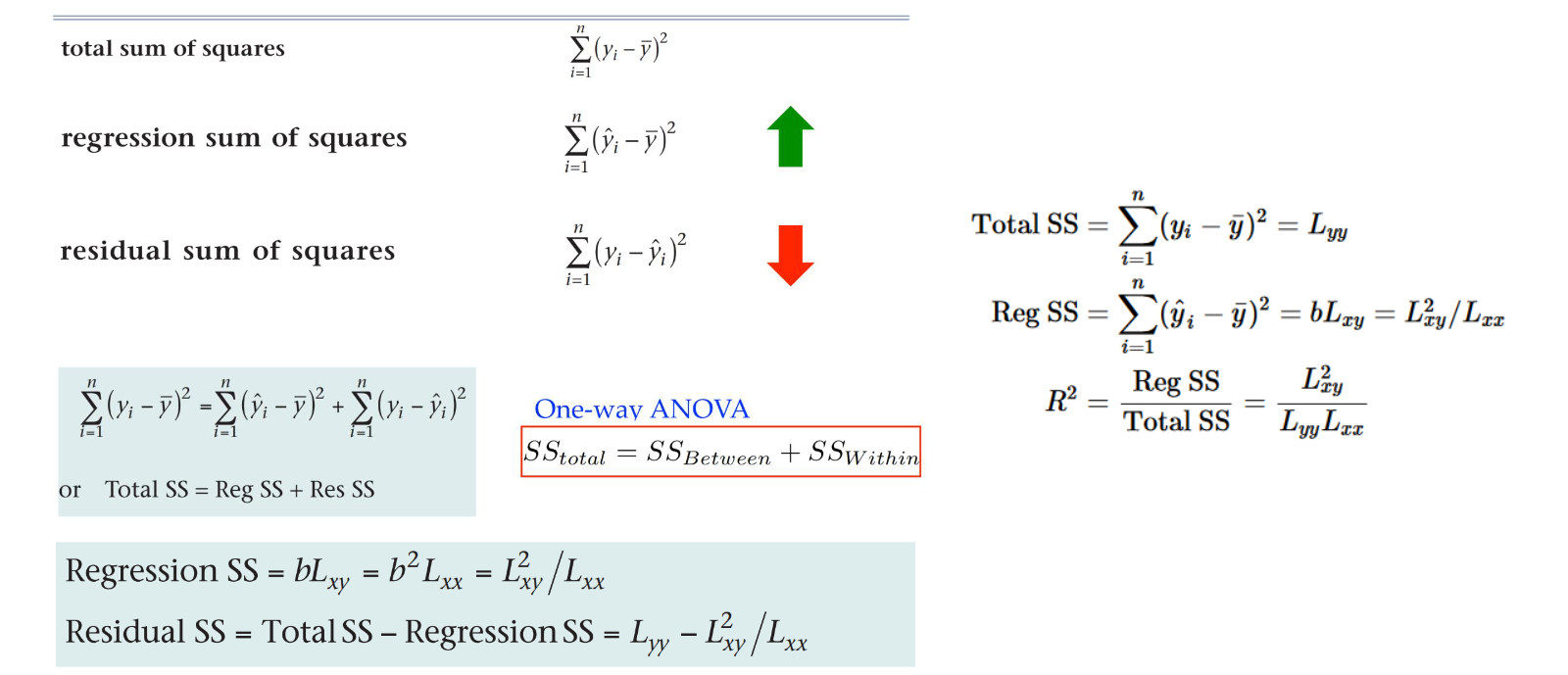
另外,还有几个很有用的变量,在线性回归和相关性分析中会被用到: $L_{xx},L_{yy},L_{xy}$ ,这三个变量反映了自变量x、因变量y的误差平方和,以及x与y的交叉项的误差平方和,它们的计算公式如下图所示:

R语言中,线性回归的函数接口是 lm(formula,data) ,其既可以做一元线性回归,又可以做多元线性回归。
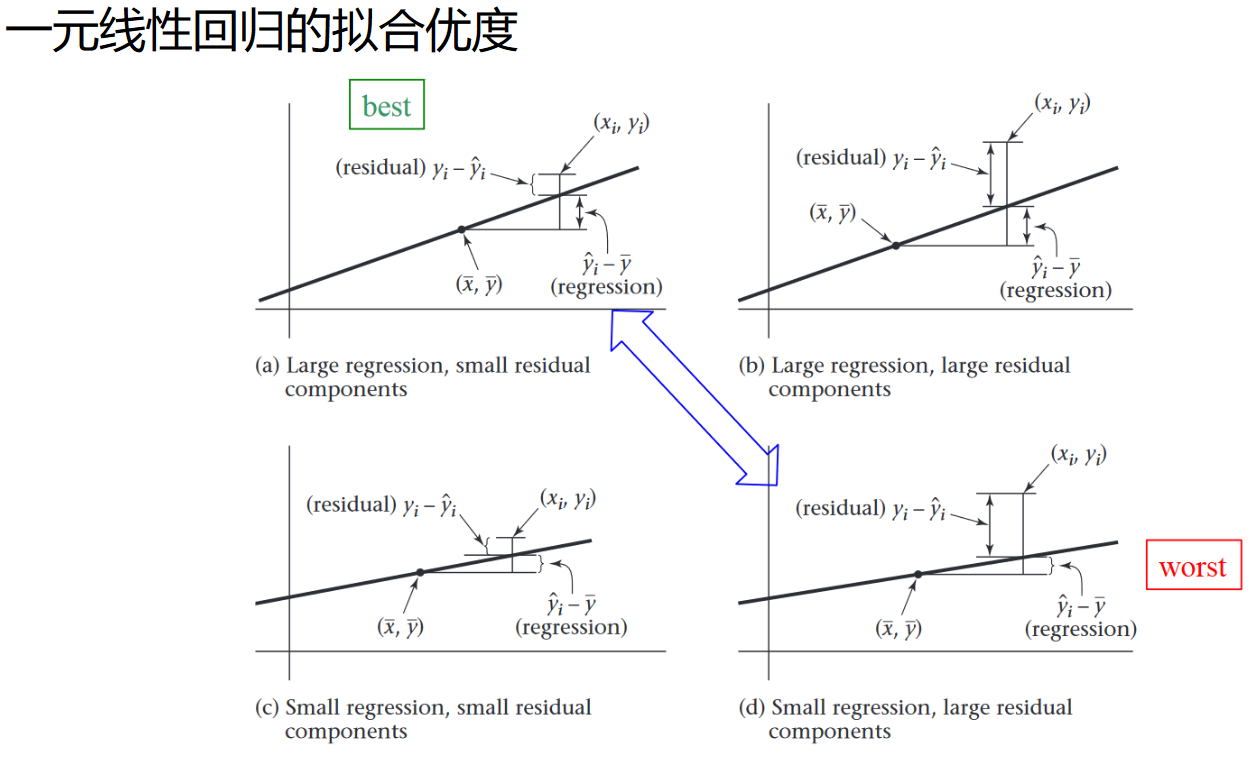
拟合优度
拟合优度是一个判断线性回归好与坏的指标,其衡量了回归模型对变量方差的解释程度(RegSS)在变量的总方差(TotalSS)中的占比, 其计算公式( $R^2$ )如下:
一元线性回归的F检验和t检验
F检验看的是所有自变量对因变量的解释程度,其原理类似ANOVA的F检验。

t检验看的是单个自变量对模型因变量的解释程度。对于一元线性回归来说,t检验和F检验是等价的检验过程;但是对于多元回归模型来说,F检验看的是一整个模型的显著性,而t检验看的就是每个自变量各自的显著性了。

(二)相关性系数
常用的相关性系数有两个:Pearson相关性系数(检验变量之间的线性相关性)和Spearman相关性系数(检验非线性的相关性)
Pearson相关性系数
Pearson相关性系数的定义有两个,分别是基于协方差的定义与基于一元线性回归方程 $R^2$ 的定义。注意存在关系式 $r=\sqrt{R^2}$ 。Pearson相关性系数的取值范围是 [-1,1] ,当r=-1时代表显著的负相关,r=1代表显著的正相关。

Spearman相关性系数
Spearman 相关系数衡量的是两列数据的排名之间的线性关系,其先将两组数据的值分别转换为各自的秩次排名(rank),然后对rank做Pearson相关性系数的计算。另外,存在一个简化公式 $\rho=1-\frac{6\sum{d_i^2}}{n(n^2-1)}$ ,在数据中没有秩次并列(“结”,tied ranks)时可以用于简化计算。

二、多元线性回归
最小二乘法公式推导:

显著性检验(F检验):

另外,R语言中有函数接口 anova() 可以用来比较两个线性回归模型。下面以R语言内置数据集 BreastCancer 为例,展示模型比较的过程:
1 | df3.omit <- na.omit(BreastCancer) |
output:
1 | ## Analysis of Deviance Table |
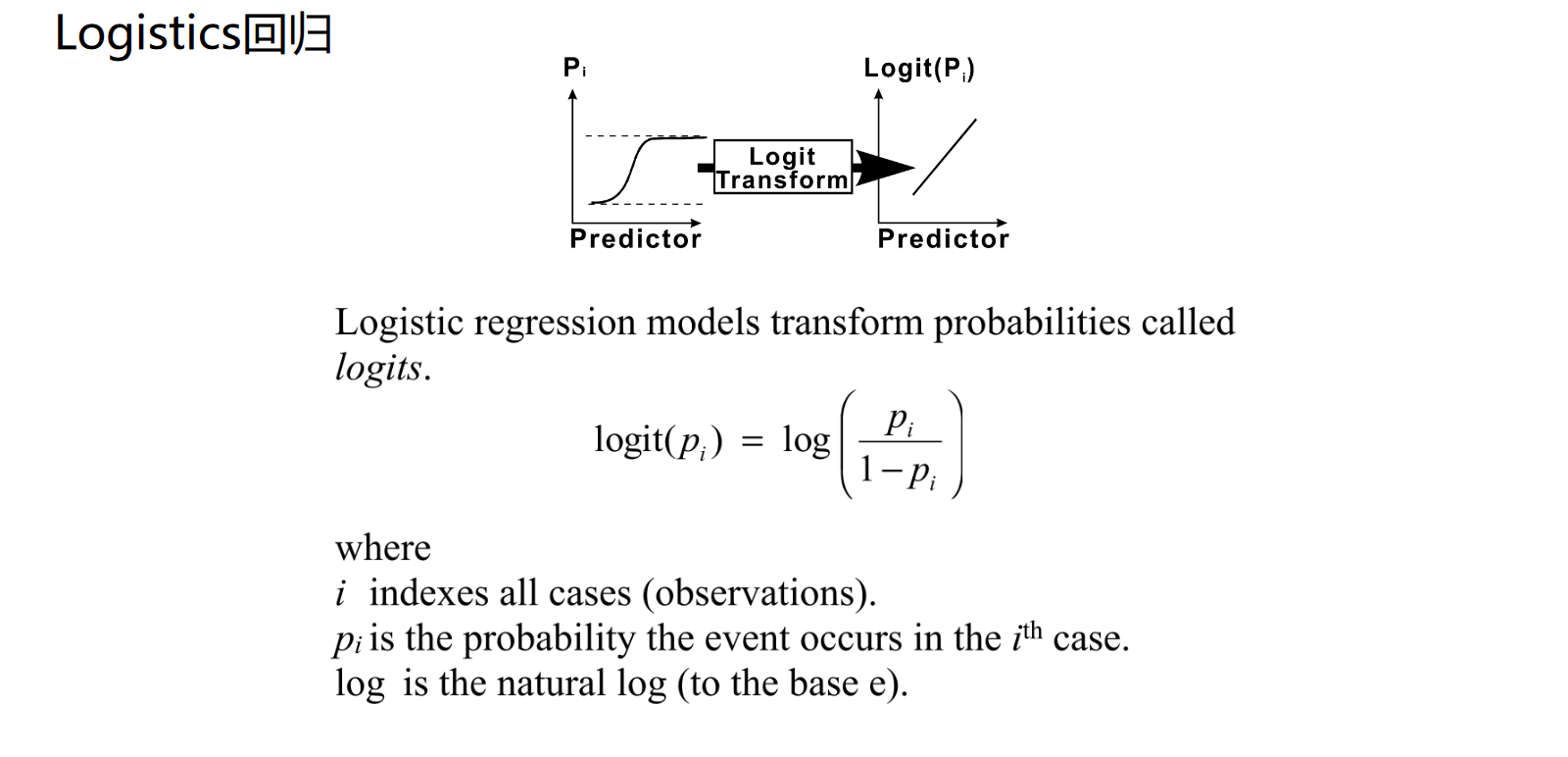
非线性回归与逻辑回归
非线性回归:使用一些函数变换,可以将模型变为线性回归。

逻辑回归:其使用 logit() 函数进行变换,值域为 (0,1) ,常用于机器学习模型的激活函数当中。