PubMed API的一些探索
PubMed数据库不仅可以在网页端使用,还可以通过API在程序中调用。配合Echarts工具,可以拿来做引文网络构建;配合LLM,还可以作为文献调研过程中的智能助手。
下面是近期的一些探索。
一、NCBI API介绍
参考:
PubMed是NCBI数据库下属的一个医学类研究论文的数据库,其中存储了与论文有关的许多信息,包括文章摘要、引文信息等。
作为世界上最大的生物学研究综合性数据库之一,NCBI提供了一组叫做Entrez Programming Utilities的工具,以帮助开发者通过程序访问数据库中的内容。这组工具包含了许多命令行实用程序,也包含一些网页API。而我们今天要使用的,就是其中的efetch和elink这两个网页API接口。
(一)准备工作:API key的申请
为了能够正常使用NCBI的API,我们首先需要申请一个API key。当不使用API key时,NCBI数据库的检索工具(E-utils)允许用户最多每秒提交3次请求,否则返回error;反之,可以达到10次每秒。
首先我们需要去NCBI账户页登录账户,此处可以使用Google Account、 ORCID、 Microsoft等多种第三方账户登录,也可以注册一个NCBI账户并登录。

登录后,在账户页右上角点击账户按钮,会弹出如下的菜单栏。在这个菜单栏里,选择Account setting进入账户设置页面。

在账户设置页面的最下方,会有一个API Key Management的栏目,在此处可以生成并管理我们的API key。新用户没有API key,可以点击按钮生成。每个用户只能拥有一个API key,如果旧API key丢失或泄露,可以在已有API key栏目中点击Replace重置。

记住这段API key,后面我们需要用。
(二) Efetch:获取给定PMID文章的详细信息
https://www.ncbi.nlm.nih.gov/books/NBK25499/#chapter4.EFetch
Efetch API的URL是 https://eutils.ncbi.nlm.nih.gov/entrez/eutils/efetch.fcgi,其允许通过GET方式进行数据请求(即,在URL之后使用问号”?“连接参数,参数以键值对的格式给出)。这一API接受的参数包括db(要检索的数据库,此处为pubmed)、id(数据库中的条目ID,此处为PMID)以及api_key。
一个示例URL如下:
1 | https://eutils.ncbi.nlm.nih.gov/entrez/eutils/efetch.fcgi?db=pubmed&api_key=ae4e7d262dc452dece0be6c4a7e06d9ccc09&id=37379837 |
上面这段URL是对pubmed请求了一篇PMID为37379837的研究论文(Huang, Jiaying et al. “Discovery of deaminase functions by structure-based protein clustering.” Cell vol. 186,15 (2023): 3182-3195.e14. doi:10.1016/j.cell.2023.05.041 )。API的返回值是一份包含了这篇论文相关信息的xml文档,如下图所示。xml中包含了文章标题、发表时间、作者列表、文章摘要、引用文献等信息,可以使用程序进行进一步的处理。

Efetch的API允许同时请求多篇PMID文献的数据,各PMID之间需要以英文逗号隔开。例如:
1 | https://eutils.ncbi.nlm.nih.gov/entrez/eutils/efetch.fcgi?db=pubmed&api_key=ae4e7d262dc452dece0be6c4a7e06d9ccc09&id=24651067,32286628,32184769,31606751 |
上述URL同时请求了24651067,32286628,32184769,31606751这4篇文章。返回的xml结构大致如下(为了节约篇幅,折叠了xml中的一些节点)。可以看到,这五篇文章分别存储在4个<PubmedArticle>标签当中,我们可以对这4个<PubmedArticle>标签分别解析,以获得这些文章的信息。考虑到XML的解析速度远快于网络请求的速度,因此如果要查询的文献较多,将他们放在一起仅做一次网页请求,是一种比较不错的提速方法。

(三)Elink:获取PMID文章之间的联系
Elink API的URL是 https://eutils.ncbi.nlm.nih.gov/entrez/eutils/elink.fcgi,同样的,其允许通过GET方式进行数据请求。这一API接受的参数包括db(要检索的数据库,此处为pubmed)、id(数据库中的条目ID,此处为PMID)、api_key、cmd(排序模式等)。
一个示例URL如下:
1 | https://eutils.ncbi.nlm.nih.gov/entrez/eutils/elink.fcgi?db=pubmed&api_key=ae4e7d262dc452dece0be6c4a7e06d9ccc09&id=37379837 |
返回的xml文档如下:

这个xml中将文献大致分为了下面这几类,不同类别之间存在交集:
- pubmed_pubmed: 表示与查询文章相关的文章,这些文章可能在内容、主题或研究方法上有相关性。
- pubmed_pubmed_alsoviewed: 表示查看了查询文章的用户也查看了这些文章,这些文章可能具有相似的主题或读者兴趣。
- pubmed_pubmed_citedin: 表示引用了查询文章的文献,这些文章直接引用了我们检索的那一篇文章。
- pubmed_pubmed_combined: 结合了多种关系类型的链接,例如引用、共同被引用等。
- pubmed_pubmed_five: 表示与查询文章相关的前五篇文章,按相关度排序。
- pubmed_pubmed_reviews: 表示与查询文章相关的综述文章。
- pubmed_pubmed_reviews_five: 表示与查询文章相关的前五篇综述文章,按相关度排序。
如果我们只关注文章的引文信息的话,仅需要解析 pubmed_pubmed_citedin 这一类别列出的文献即可。
二、citationMap工具的打造
最初令我关注到这个API的动力是希望创建一款可视化引文网络的工具,以厘清文献调研的文章是否有共同引用或共同被引。如前所述,Efetch和Elink这两个API提供了大量信息,足够我们构建引文网络了。但是还有两个问题需要解决:如何从API返回的xml中提取信息,以及,如何将这些信息进行可视化。
python自带了XML标记语言处理模块,我们只需要在程序中简单的 import xml.dom.minidom 即可利用这一模块提供的xml解析能力。因此,第一个问题能够很方便的解决。
然而,python自带的数据可视化工具并没有特别好用。我们希望引文网络工具可以提供一定的交互能力,如拖动节点、查看节点信息等,matplotlib做不到这些。好在开源社区提供了一个叫做Echarts的网页端数据可视化工具,它提供了强大的交互功能。(说起来,这个工具最初是百度开发的,后面百度捐赠给Apache社区了;感谢百度在开源领域做出的贡献)
我们这一工具的打造分两步:XML文档的解析,以及基于Echarts的可视化。
(一)XML文档解析
这一部分涉及的代码量很大,为了节约篇幅,此处只介绍大概的原理。
在一切的开始,我们需要进行网页请求,以获取xml文档。下面给出了网页请求的函数,其接受一个PMID列表的输入,使用这个PMID list构造URL并请求API,最后返回xml文档的内容。
1 | import requests |
示例输出如下:

对这段xml的解析需要用到python自带的XML库。如下代码块所示,使用parseString().documentElement可以将xml文档编程一个xml document对象树,进而可以使用面向对象的方法对文档中的各个节点进行访问。例如,使用getElementsByTagName("PubmedArticle")可以把xml中所有标签为PubmedArticle的节点提取出来,并构造为一个列表。
1 | from xml.dom.minidom import parseString |
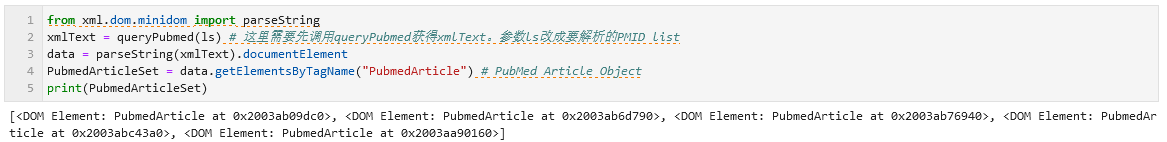
接下来,就是漫长而无聊的对这棵对象树解析过程了。为了展示这个过程有多无聊,下面给出了获取第一篇文献的PMID的代码,其中需要反复用到getElementsByTagName。

Efetch文档和Elink文档的整体解析过程此处省略。总之,我们完成了对xml文档树的解析过程,获得了与文献及其引文网络有关的大量信息。下面我们再来看一看如何使用Echart做可视化。
(二)基于Echarts的可视化
如题。我们要展示文献之间的引用和被引关系,因此需要一类能够显示节点和网络的图表类型。幸运的是,Echarts中提供了一类关系图表(如下图),可以满足我们的要求。

citationMap这个工具的设计思路大致是这样的:
- 用户提供一些文献作为初始文献,我们从reference(“参考文献”)和citations(“引用文献”)两个方向开展查询:
- “参考文献”: 查询这些初始文献引用了哪些历史文献。在这些历史文献中,存在一些共同引文,这些共同引文一般是领域内的经典文献,需要重点关注。
- “引用文献”: 查询有哪些新发表的文献引用了这些初始文献,这一部分文献代表了领域的发展现状。
- 不论是reference关系还是citations关系,都用一条边来表示,表明两篇文献之间存在关系。
- 以网络关系进行可视化,网络上的每个节点代表一篇文献,用不同的颜色来区分初始文献、参考文献和引用文献。用户可以点击或拖动这些节点,当点击时,侧边栏展示出文献的详细信息。
为了实现节点的点击和拖动效果,我们可以使用Echarts中提供的力引导布局方式进行节点展示。其中涉及较多的Javascript代码,限于文本篇幅此处不展开,感兴趣的读者可以在下面的网页上尝试Echarts提供的示例:
- https://echarts.apache.org/examples/zh/editor.html?c=graph-force
- https://echarts.apache.org/examples/zh/editor.html?c=graph-webkit-dep
需要注意的是,与matplotlib、ggplot或者matlab等不同,Echarts进行数据可视化展示,需要提供的数据源自json文档,包括各个点的坐标、颜色等属性都需要在json中进行描述(这一部分的逻辑已经在python中实现了)。
citationMap的另一项需求是点击节点展示信息。幸运的是,Echarts为鼠标点击事件提供了API接口,我们只需要捕获鼠标点击事件发生时的param参数并正确解析即可。下面是这一部分的处理逻辑(需要配合html才能实现正确的效果)
1 | // 下面的代码用于响应鼠标点击动作,使用`params`参数可以获得当前节点的所有信息 |
具体到我们的citationMap这个工具上,可视化部分需要分两部分进行:
- python程序部分:用来解析xml文档和生成json文件,后者是Echarts进行可视化展示所必需的
- html页面:用来加载json并调用Echarts展示网络图。
(三)工具成品以及使用方法
citationMap这一工具已经开发完成,我将其上传到了Github存储库 - citationMap当中。
在这一工具中,核心的代码文件主要有两个: app.py 和 index.html 。前者接受用户输入,解析xml文档和生成json文件,后者加载json并调用Echarts展示网络图。为了方便用户使用,在app.py主函数中会开启一个web服务器的进程,并调用浏览器打开index.html以展示结果。
1 | usage: app.py [-h] [-f FILE | -l LIST] [-m MAX] [-p PORT] |
如上所示,这一工具目前暂时只支持命令行调用,有两种使用模式: -f 文件输入模式和 -l 命令行传参模式。不论哪一种模式,用户的输入都只能是一系列文章的PMID。
另外,-m 参数用来调整可供解析的文献数量上限(为了确保数据可视化时的性能,默认只解析十篇文章;如果需要解析更多篇文章的话,可以加这个参数手动调整); -p 参数用来设置web服务器的端口号(默认是随机端口)。
一个使用示例如下(命令行传参模式):
- 1、打开终端,切换到citationMap所在目录
- 2、运行下面这行指令
1 | python app.py -l 24651067,32286628,32184769,31606751 |
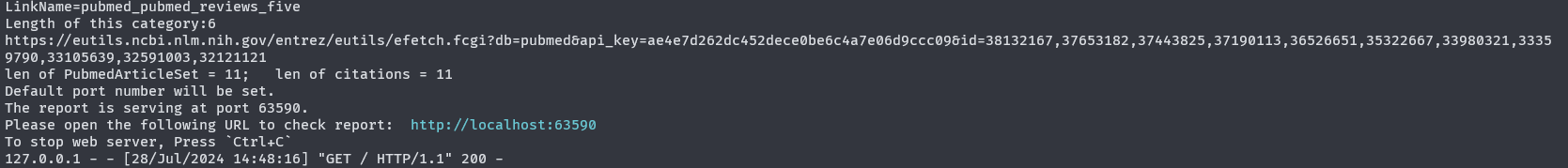
- 3、此时,程序会开始工作,屏幕上会闪过一些信息,表明文档解析的进程(如下图所示)

- 4、当解析完成后,在当前目录下会生成一个
citation_map.json文件。同时,python程序也会启动web服务器进程(终端会输出如下信息,以蓝色字体输出URL)。如果用户的系统是Windows或者macOS,则会直接启动系统浏览器并打开这一URL
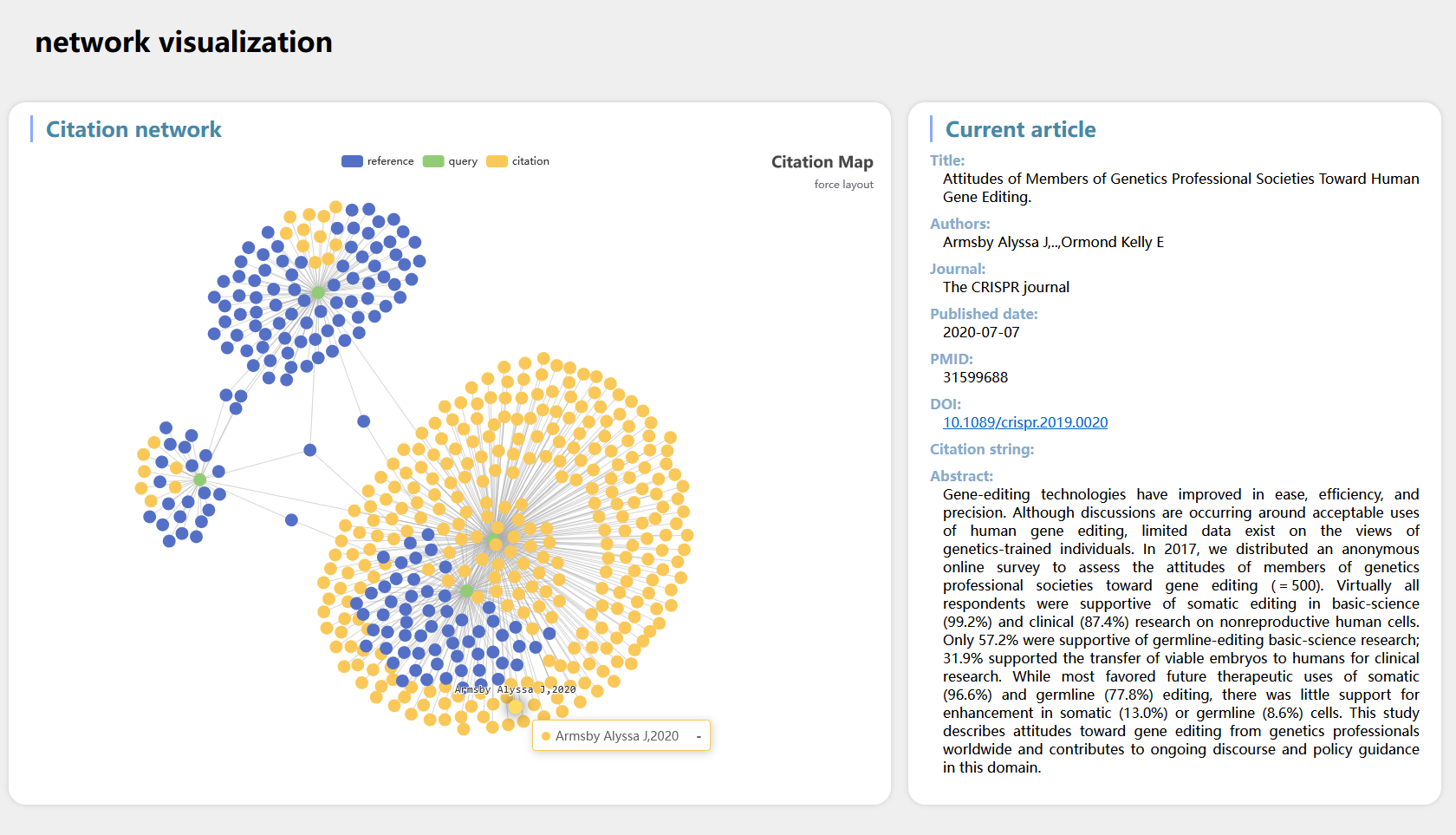
- 5、之后的操作在浏览器中进行。我们将看到类似下面的网页内容,其中绿点代表初始文献,蓝点代表参考文献,黄点代表引用文献。鼠标悬停在节点上时会弹出一个简略的提示框,包含第一作者和发表年份信息;点击节点,则会在右侧面板中展示出这篇文章更具体的信息,包括标题、摘要、DOI等。
文件传参模式与之类似,只不过PMID列表是存储在一个文本文件中的(一行一个PMID),使用-f <file path> 参数进行传参,此处不再赘述。
(四)未来计划(aka.画大饼)
目前这个工具的使用方法还比较繁杂,需要在命令行界面下启动app.py主程序,并在浏览器中进行结果的检查和浏览。鉴于python强大的服务器后端处理能力,未来将对citationMap网站化,用户只需要在网站输入框里输入一系列的PMID(就像百度或者Google那样),就可以一键获得citationMap的信息。
另外,目前的交互依旧比较简陋,只能查看引文网络和获取有限的文章信息。未来将进一步拓展网页的功能,提供诸如节点过滤、二次查询、结果导出等功能,以更进一步的方便使用。
鉴于pubmed数据库自身的一些bug,部分文献的引文信息不全,因此之后还会考虑将其他文献数据库纳入检索范围之中。
(几张饼先画下了,啥时候实现不确定,手动狗头)
三、当PubMed API遇上大模型
这一部分的内容算是上述工作的副产品。某天查文献的时候想到,既然LLM+实时网页搜索就能获得那么多有用的信息,那么LLM+PubMed API是不是也是如此呢?
于是我对上面的代码进行了改造:首先,去除了查询reference和citations文献的处理过程,仅使用Efetch获得的文献信息进行后续的操作。其次,得到的结果储存在一个json字符串中,后者用于喂给大模型。
1 | # 接着上述那个app.py,加两个新的function: |
喂给大模型:

(这里我导入了之前用过的一个通义千问大模型API,所以直接chat()就可以。如果没有大模型API的话,也可以把 PROMPT_text 打印出来,然后复制粘贴到大模型的聊天框里面)
后记
citationMap这个工具其实从去年年底就开始开发了(见 《更新日志(2023-12-17)》 ),中间因为各种事情拖延了大半年,直到现在这个工具才算比较完善。
API+LLM的部分,其实也是前段时间摸鱼工作搞出来的探索,只不过没有太深入。总的来说,目前各个大模型的智能程度足以进行一些简单的文献调研工作,虽然要做得更精细还得靠我们自己亲自阅读。
又是一年七月末,暑气已至而假期未到。昨天看了巴黎奥运会的开幕式,火炬手cos《刺客信条》的创意以及穿插其中各种表演属实惊艳我了一把。不知道说些什么结尾,就祝大家夏天快乐吧 ~ 愿读者朋友们生活如夏花般绚烂精彩。
以上。