使用ggtree绘制系统发育树
绘制系统发育树的方法
一、基本概念:nwk格式文件
nwk格式是一种存储系统发育树的格式,更具体的信息可以参考https://evolution.genetics.washington.edu/phylip/newicktree.html。在这一类型的文件中,系统发育树的节点和分支由圆括号的层次关系所表示。
一颗系统发育树的表达式以分号结尾,用圆括号和逗号标记节点和分支。节点名称可以为空,也可以是一串可打印字符,但是不能包含空格、逗号、分号、圆括号和方括号。
一种拓展的表示方法是在节点名称后加分支长度,二者以冒号隔开。例如
1 | (B:6.0,(A:5.0,C:3.0,E:4.0):5.0,D:11.0); |
另外,由于分支和节点除了拓扑关系以外,不存在其他的排序问题,因此对于同一棵数,可以有多种表示方法。例如,下面五条表达式,所表达的是同一棵树。
1 | (A,(B,C),D); |
二、使用ggtree绘制系统发育树
ggtree是一个基于ggplot的R包,可以在R中对系统发育树进行可视化。
具体使用说明可以参考bioconductor上的主页和treedata-book文档进行了解。
安装方法:
1 | BiocManager::install("ggtree") |
一个简单的例子:
1 | library("ggtree") |
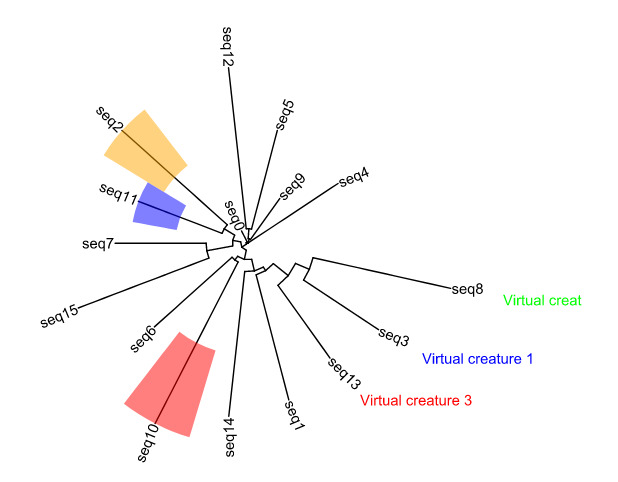
上面的代码读取了ggtree_test.tree这个文件,并使用ggtree()这个函数实现了可视化。为了后期修图方便,此处我们将其保存为svg矢量图格式。得到的结果如下图所示。
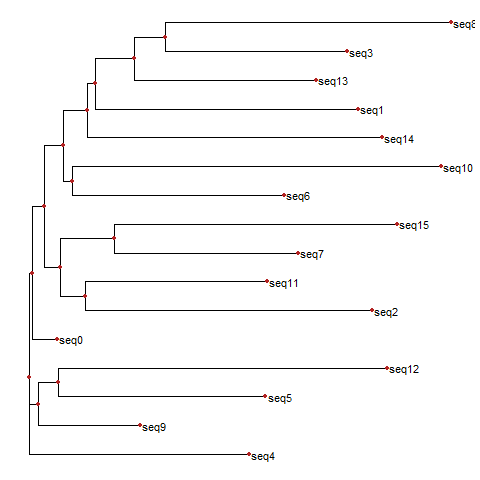
ggtree包依赖于ggplot,因此一些针对ggplot的图片参数调整方法同样适用于ggtree。例如,我们可以通过下面的代码,得到一棵更加美观的树。
1 | myfig2 <- ggtree(mytree,layout = "circular")+ |