如题。先前在朋友的博客中看到了一个利用大模型进行文献调研的研究(参见 《文献pdf改名&AI消化》 )。正好这一阵子有文献调研的需求,于是在此基础上进行了一些更深入的探索。
本文主要分为三个部分:①利用网络爬虫获取PubMed的论文全文内容;②通过prompt工程调用大模型,以json文档的形式返回消化结果;③pandas批量处理保存为Excel表格。下面是探索结果
一、利用网络爬虫获取PubMed的论文全文内容
此处参考前段时间我发布在GitHub上的一个项目: pubmed_spider: A spider program for downloading text from pubmed
众所周知,Pubmed是一个很全面的医学研究论文的数据库,并且每一篇论文都有唯一确定的PMID标识符来表示,因此我们可以通过PMID获得论文的信息页面。
除此之外,Pubmed还附带了一个PMC的数据库,其中存储着论文的全文,以PMCID标识符来表示。一些受受 NIH 资助的研究成果,以及开放获取的研究论文,会被PMC数据库收录,因此当我们知道一篇文章的PMCID时,我们就可以获得论文的全文。
PMCID与PMID之间存在一对一的映射关系。当我们知道一篇论文的PMID,我们就可以通过解析PubMed的页面,获得PMCID,进而获得全文。而Pubmed的搜索支持根据论文标题进行模糊匹配,因此当我们知道论文标题,我们也可以通过解析PubMed页面获得论文PMID。这就是 pubmed_spider 这个项目的原理。
(插一句话,pubmed_spider 是用户提供一系列论文标题,通过爬虫获得txt格式的论文全文内容,其最终目的服务于大模型的知识库RAG服务)
下面是具体代码:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
| import requests
from bs4 import BeautifulSoup
headers = {'User-Agent':'Mozilla/5.0 (Windows NT 10.0; Win64; x64; rv:109.0) Gecko/20100101 Firefox/115.0'}
def extract_page_content(url):
req = requests.get(url,headers=headers)
req.encoding = "utf-8"
txt = req.text
bs4obj = BeautifulSoup(txt,'lxml')
page_content = bs4obj.get_text().strip()
return page_content
def query_from_pmc(pmcid):
url = f"https://pmc.ncbi.nlm.nih.gov/articles/{pmcid}/"
text = libgeturl.extract_page_content(url)
if(text is None): text = ""
text = text.replace("\n","")
text = text.replace("```","")
text = text.replace('"""',"")
return text
def query_from_pm(pmid):
url = f"https://pubmed.ncbi.nlm.nih.gov/{pmid}/"
text = libgeturl.extract_page_content(url)
if(text is None): text = ""
text = text.replace("\n","")
text = text.replace("```","")
text = text.replace('"""',"")
return text
|
通过PMID获取PMCID的代码逻辑稍微有些复杂,因为需要利用BeautifulSoup(bs4)库解析页面上的DOM元素。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
| def get_pmcid_by_pmid(pmid):
url = f"https://pubmed.ncbi.nlm.nih.gov/{pmid}/"
req = requests.get(url,headers=headers)
req.encoding = "utf-8"
txt = req.text
bs4obj = BeautifulSoup(txt,'lxml')
pmcid_list = []
all_ul_list=bs4obj.find_all("ul", class_=['identifiers'])
for li in all_ul_list:
all_link_list = li.find_all("a")
if(len(all_link_list)>0):
for link in all_link_list:
attrs = link.attrs
if("data-ga-action" in attrs):
if("PMCID" in attrs["data-ga-action"]):
pmcid = link.get_text().strip()
if(pmcid not in pmcid_list):
pmcid_list.append(pmcid)
break
return pmcid_list
|
二、通过prompt工程调用大模型
因为文献调研任务涉及到论文中的一些关键信息的提取,我改进了朋友博客文章中的那个prompt,要求以json文档的格式返回生成结果:

1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
| 你是一位生物医学领域的文献总结大师,按照用户提出的格式总结上传的学术论文,用json输出内容。
除了json文档本身,请不要输出其他任何无关信息,包括对这篇论文或这份json文档的解释。
格式要求如下:
{"Title": "[请填写文章标题]",
"Population": "[列出文章中使用的人群队列名称或群体来源]",
"Species": "[列出文章中使用的实验动物物种,如`mus musculus` , 物种名尽可能使用拉丁名,如果没有合适的拉丁名就使用文献中使用的物种名原名]",
"Type": "[归纳一下文章的研究属于哪一种类型,如 `review`, `new algorithm`,`new technology`,`biological research`,`data source`等]",
"Method": "[列出论文中使用的群体遗传学方法或正选择压力检测方法。如果没有则以字符串None填充这个条目]",
"Tissues": "[列出论文中的研究涉及了哪些组织或细胞系,如`Liver`,`Brain`等]",
"Data": "[列出论文的数据可访问性部分给出的数据访问链接,或GEO等数据库的访问号。如果没有则以字符串None填充这个条目]",
"Code": "[列出论文的数据可访问性部分给出的代码链接,如Github存储库地址。如果没有则以字符串None填充这个条目]",
"Contribute": "[用简洁的语言总结论文的主要贡献]",
"Significance": "[说明这项研究对学术界或实际应用的意义]",
"Suggestion": "[列出作者提出的未来研究方向或建议]"
}
下面是论文的标题和摘要页:
(-abstract-)
下面是论文的正文:
(-content-)
|
prompt中的 (-abstract-) 字段和 (-content-) 字段,需要在实际调用大模型API时替换为论文的摘要内容( query_from_pm )和论文全文( query_from_pmc ) 。
上面的json格式模板仅作为一个示例,用于群体遗传学的调研 (因此, Population, Species, Method, Data, Code这几个字段对我们来说很重要)。在处理其他类型研究时,可以根据需要修改json的字段内容。
三、pandas批量处理保存为Excel表格
假设我们已经下载好了所有论文的PMID信息和标题信息(例如,通过PubMed检索页面导出),并保存为文件 csv-articles-set.csv 。下面的代码展示了如何进行批量处理:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
| import pandas as pd
import libqwen
article_df = pd.read_csv("csv-articles-set.csv")
res_df = pd.DataFrame()
error_msg_dt = {}
for i in range(article_df.shape[0]):
print(i)
record = article_df.iloc[i,:]
pmid = record["PMID"]
pmcid = record["PMCID"]
text_ab = query_from_pm(pmid)
text_ct = query_from_pmc(pmcid)
message = prompt
message = message.replace("(-abstract-)",text_ab)
message = message.replace("(-content-)",text_ct)
libqwen.hist_msg = []
res = libqwen.chat(message)
res1 = res.replace("```json","").replace("```","").strip()
try:
dt1 = json.loads(res1)
df1 = pd.DataFrame(dt1,index=[0])
res_df = pd.concat([res_df,df1],axis=0)
res_df = res_df.reset_index(drop=True)
res_df.to_parquet("LLM-process-result.parquet", compression="brotli")
except Exception as e:
error_msg_dt[i] = e
pass
df1 = pd.read_parquet("LLM-process-result.parquet")
df_merge_and_combined = article_df[["PMID","Title","Publication Year","Journal/Book","DOI"]]
df_merge_and_combined.columns = ["PMID","Title-1","Publication Year","Journal/Book","DOI"]
df_merge_and_combined = df_merge_and_combined.join(df1)
df_merge_and_combined.to_excel("LLM-process-result.xlsx")
|
其中,libqwen.py 的内容如下(在之前的LLM命令行聊天软件的基础上改的,因此显得很凑合。主要是需要这里的 chat() 函数接口,通过这个接口获得LLM处理的返回值)
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
| import requests,json,datetime,os,sys,platform
API_KEY = "sk-82b3bfa35dce4e93877d7a54841d157d"
headers = {
'Content-Type': 'application/json',
'Accept' : 'application/json',
'User-Agent':'Mozilla/5.0 (Windows NT 10.0; Win64; x64; rv:109.0) Gecko/20100101 Firefox/115.0',
'Authorization': f"Bearer {API_KEY}"
}
def get_response_with_stream(messages):
data_payload_dt = {
"model":'qwen-long',
"stream":True,
"messages":messages
}
payload = json.dumps(data_payload_dt)
url = "https://dashscope.aliyuncs.com/compatible-mode/v1/chat/completions"
response = requests.request("POST", url, headers=headers, data=payload, stream=True)
full_content = ""
for line in response.iter_lines():
txt = line.decode("UTF-8")
txt = txt.strip()
left_trunc_pos = txt.find('{')
left_trunc_pos = left_trunc_pos if(left_trunc_pos>0) else 0
txt = txt[left_trunc_pos:]
if(txt=="data: [DONE]"): continue
try:
obj = json.loads(txt)
response_msg = obj["choices"][0]["delta"]["content"]
full_content += response_msg
except:
full_content += txt
return full_content
hist_msg = []
def chat(msg):
global hist_msg
hist_msg.append({"role":"user","content":msg})
message = get_response_with_stream(hist_msg)
if(len(message)==0): message="I don't understand this question."
hist_msg.append({"role":"assistant","content":message})
return message
|
四、效果展示与注意事项
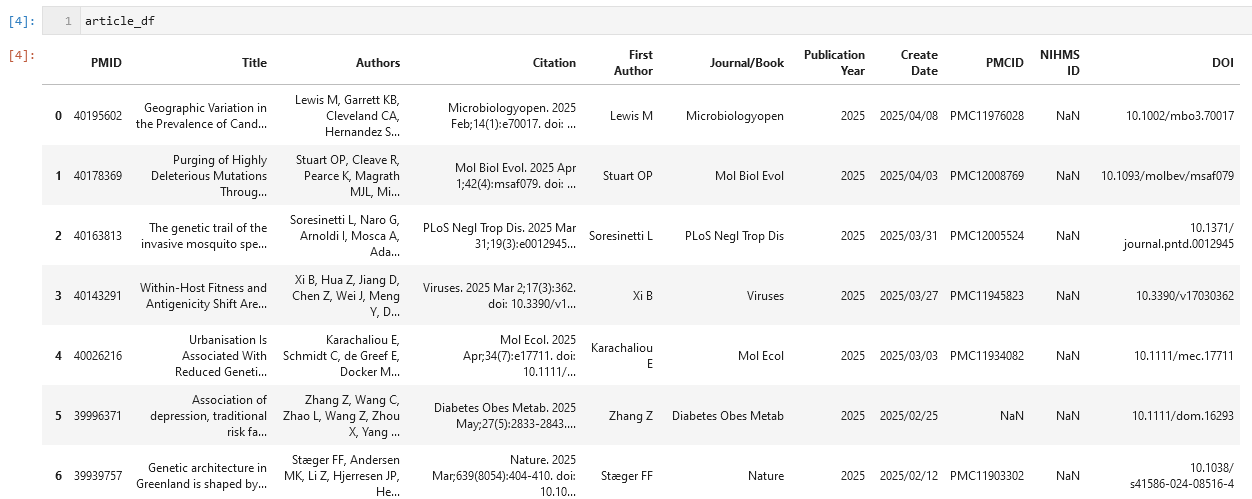
我们以一个小文献集为例,这一批文献是从PubMed上以 “Population genetics” 为关键词检索到的,部分内容如下:
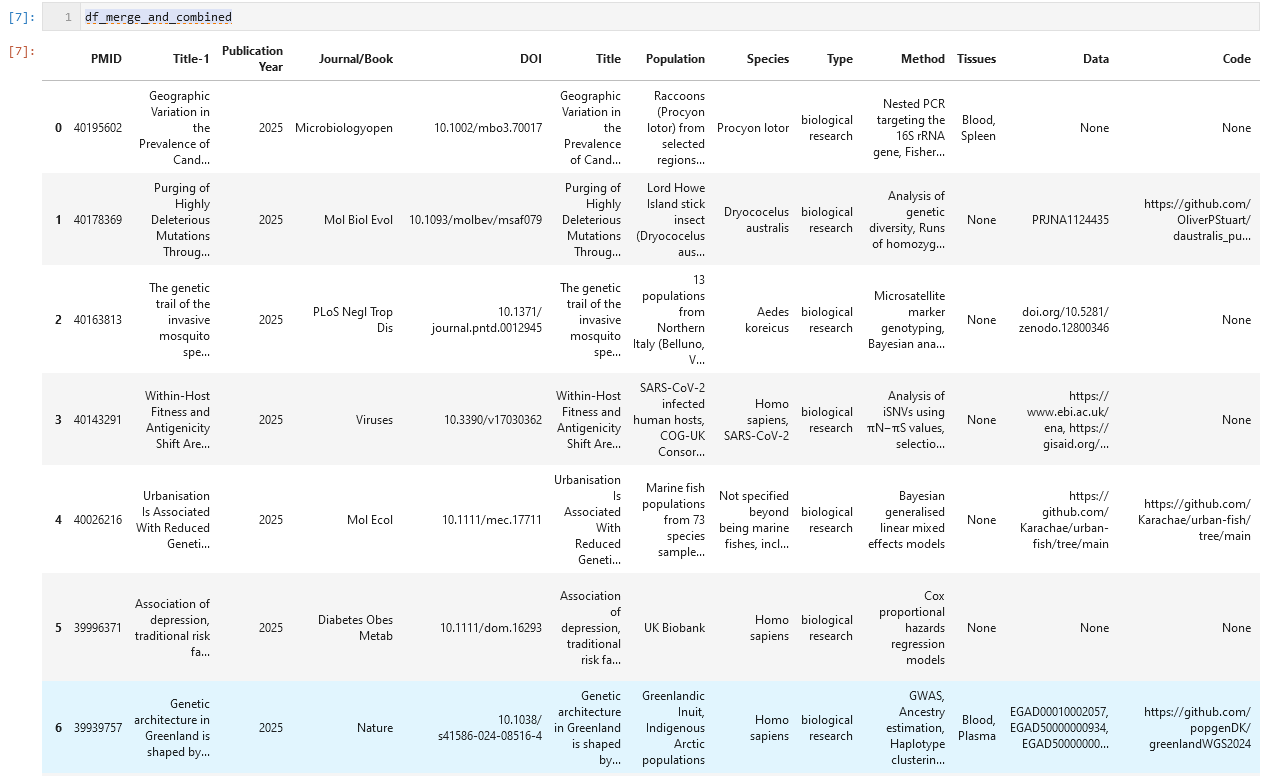
通过上面的大模型消化,我们最终得到的成品如下表所示,其中的 Populations, Species, Data, Code等列是我们所关心的。
另外,关于LLM处理的准确性问题,经过实测,qwen-long的准确性还是比较好的。同期我也测试过百度ERNIE的系列模型,发现模型的幻觉问题比较严重,因此不建议使用ERNIE系列的模型。如果需要更准确的处理结果,大家也可以切换其他模型API,例如deepseek、gemini等。另外,这一工具在文献初筛的时候用比较好,筛选完以后最好还是得再自己精读一下比较稳妥。
以上。


